Le projet Human Brain étudie le cerveau et ses pathologies. La première approche consiste à reproduire un modèle réaliste du cerveau, basé sur des données neuroscientifiques et génétiques détaillées. D’autre part, de grandes quantités de données concernant de nombreux aspects d’une maladie doivent être collectées dans un format de données unique. L’objectif est de combiner ces deux approches afin de lier les causes génétiques et neurophysiologiques complexes des maladies psychiatriques aux phénotypes complexes qui les accompagnent.
Depuis longtemps déjà, il est admis que les maladies psychiatriques telles que la schizophrénie et la dépression ne peuvent pas être expliquées de manière monocausale, mais que des approches multifactorielles sont nécessaires. Au niveau de la méthodologie de recherche, cela signifie que les approches de recherche isolées ne sont pas suffisantes, mais qu’il faut des méthodes capables d’appréhender la combinaison de mécanismes multifactoriels (par ex. au niveau génétique, neurobiologique, phénoménologique). Il en va de même pour l’étude du cerveau humain, avec ses milliards de neurones et de connexions neuronales.

L’objectif du projet Human Brain est de pouvoir explorer la complexité du cerveau et de ses maladies. Deux approches sont prévues à cet effet : D’une part, un modèle numérique du cerveau est reconstruit sur la base de données neuroscientifiques et génétiques. D’autre part, de grandes quantités de données concernant de nombreux aspects d’une maladie doivent être rassemblées dans un format de données unique. La vision de ce projet est de combiner ces deux approches et de trouver de nouvelles explications en intégrant les caractéristiques génétiques et neurophysiologiques des maladies psychiatriques et les phénotypes complexes qui y sont associés. En résumé, il s’agit donc d’une approche de la collecte et de l’analyse de grandes quantités de données relatives au cerveau humain.
“Approche ascendante” et “approche descendante”.
Dans l’approche ascendante, l’objectif de reproduire entièrement le cerveau, c’est-à-dire de le numériser, est central. Sur le plan méthodologique, il s’agit d’un projet d’intégration de données comparable au développement d’un atlas numérique basé sur différentes sources de données à différentes échelles. Un modèle numérique de la structure et du fonctionnement du cerveau est conçu sur la base des données du “Blue Brain Project” [1]. L’approche descendante consiste à collecter des données relatives aux différentes caractéristiques d’une maladie (niveau génétique et moléculaire, niveau comportemental, etc.). Ces données sont collectées dans les cliniques et les universités partenaires, restent sur les ordinateurs des institutions concernées et sont analysées par un logiciel spécial. Les paramètres de ces analyses sont ensuite stockés dans une plate-forme neuroinformatique centrale. De là, ces données sont transmises à d’autres plates-formes :
- Plate-forme de simulation (= examen du cerveau sain)
- Plate-forme dédiée aux maladies

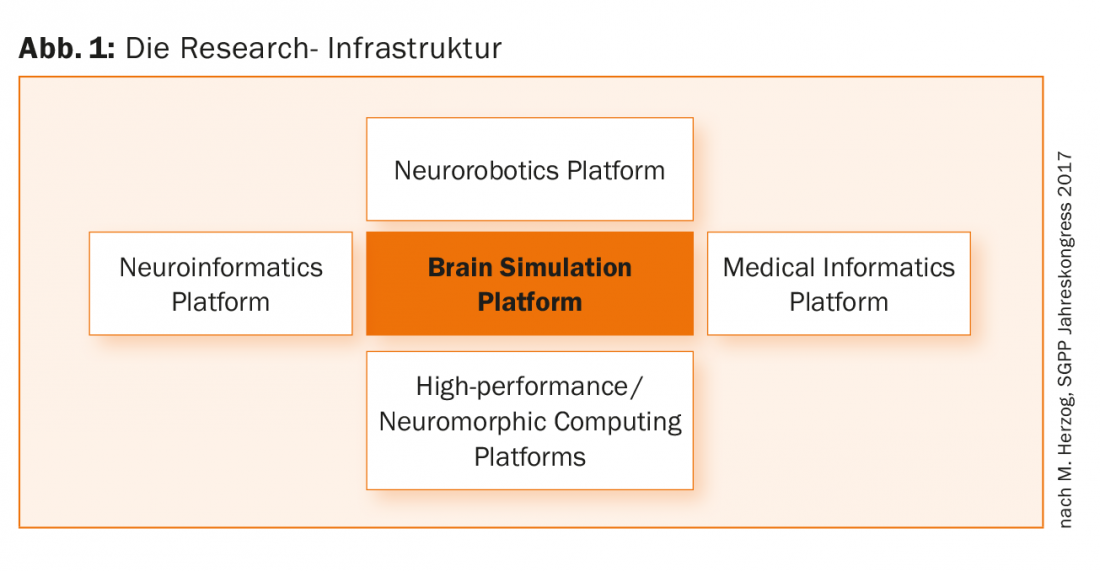
Il existe douze sous-projets au sein du projet Human Brain. Dans le cadre du sous-projet 10 (“Neurorobotics”), les scientifiques et autres spécialistes disposent par exemple d’un nouvel environnement basé sur le web (“Neurobiotics Plattform”) (fig. 1), qui offre la possibilité de combiner des modèles cérébraux avec des simulations détaillées de robots et de leurs environnements, et d’utiliser les systèmes de neurorobotique qui en résultent pour des expériences in silico [2].
La plate-forme d’informatique médicale
Le sous-projet de recherche sur les maladies mentales (“The Medical Informatics Platform”) (Fig. 1) vise à mieux comprendre les maladies mentales. Il implique 14 institutions partenaires et 5 cliniques, dont un groupe de recherche dirigé par Ferah Kherif et Bogdan Draganski, tous deux chercheurs et enseignants à l’Université de Lausanne.
Les sciences du cerveau et le diagnostic clinique doivent être reliés et les données doivent être standardisées. L’objectif est de mieux prédire et expliquer les maladies complexes en intégrant des données sur les caractéristiques cliniques (phénoménologie), étiologiques et biologiques et en les comparant au modèle numérique du cerveau.
Comment cela est-il mis en œuvre ? On mesure toutes les informations disponibles sur une maladie donnée au niveau génétique, moléculaire ou comportemental. Des algorithmes d’apprentissage automatique sont utilisés pour analyser ces données. Le réseau apprend ainsi à trouver des différences et à former des clusters au sein d’un vaste ensemble de données.
Par exemple, une analyse réalisée avec cette méthode sur un grand ensemble de données de patients atteints de la maladie d’Alzheimer [3], comprenant des données sur les caractéristiques génétiques, moléculaires et cognitives, a permis d’identifier différents sous-groupes. Cette méthode permet d’étudier comment certains mécanismes produisent une phénoménologie particulière. Dans une prochaine étape, ces connaissances pourraient être utilisées pour développer des médicaments spécifiques à des sous-groupes. Étant donné que le projet Human Brain est une infrastructure de “big data” dont le but est d’accéder à une grande quantité de données cliniques, des questions éthiques, telles que les conditions d’un “consentement éclairé”, doivent également être résolues [4].
Exemple d’application concrète : modèle d’explication de la schizophrénie
Des études empiriques montrent que les maladies schizophréniques présentent des anomalies à différents niveaux dans différentes caractéristiques et que les facteurs génétiques jouent un rôle important. Par exemple, il a été démontré que le risque de développer une schizophrénie à un moment donné de sa vie (“lifetime risk”) est de 50% pour un frère ou une sœur jumeau(e) d’une personne atteinte de la maladie. Les enfants de parents malades ont un risque de 13% de développer également une schizophrénie. Des études empiriques ont montré que les patients schizophrènes sont significativement plus lents qu’un groupe témoin de personnes en bonne santé lorsqu’ils effectuent des tâches qui sollicitent fortement leur mémoire de travail, et que les schémas d’activité neuronale dans le cortex préfrontal sont différents. Une batterie de tests a révélé des anomalies dans plusieurs tests (par exemple, le Wisconsin Card Sorting Test, le Visual Masking, le Stroop, etc. Cependant, peu de corrélations ont été trouvées entre les résultats des tests individuels et les polymorphismes de nucléotides uniques et les résultats des tests cognitifs. Cela montre qu’il s’agit d’une maladie complexe.
Herzog a illustré le défi combinatoire d’une maladie multifactorielle comme la schizophrénie sous une forme simplifiée à l’aide d’une expérience de pensée : supposons qu’il y ait huit facteurs liés à la maladie et que chacun de ces facteurs ait deux variantes (par exemple “Up”, “Down”), il y a alors 256 constellations “Up”/”Down”. Chaque composant individuel ne pose pas de problème, c’est la combinaison des différentes variantes qui est déterminante. Le fait que toutes les variantes soient “Up” ou “Down” correspond à une probabilité inférieure à 1%. Cela correspond à la prévalence de la schizophrénie dans la population générale. Pour étudier plus précisément les mécanismes des maladies complexes, il est nécessaire de disposer de grandes quantités de données ainsi que de méthodes d’analyse capables de saisir la combinaison de différentes caractéristiques et de former des clusters. Il s’agit d’un exemple d’application possible de la méthodologie de recherche du projet Human Brain.
Activités de recherche prévues
En avril 2018, un appel d’offres sera lancé pour donner plus de détails sur les conditions de participation au projet Human Brain. Une possibilité est de réaliser des collectes de données dans le cadre clinique. Il est prévu qu’un logiciel d’analyse des données soit installé sur les ordinateurs de chaque clinique et que seules les métadonnées anonymes et cryptées soient exportées vers des ordinateurs extérieurs. Les données d’origine restent donc toujours sur les ordinateurs de la clinique. Les métadonnées cryptées doivent être mises à la disposition du public et accessibles aux personnes intéressées. A l’avenir, toute personne s’inscrivant au projet pourra, par exemple, connaître l’impact d’une certaine concentration de protéines sur certains autres paramètres.
- Site web The Human Brain Project
www.humanbrainproject.eu

Littérature :
- Le Blue Brain Project. Une initiative du cerveau suisse. http://bluebrain.epfl.ch
- Falotico E, et al : Connecter des cerveaux artificiels à des robots dans un cadre de simulation complet : la plateforme Neurorobotics. Front Neurorobot, 2017 25 Jan ; https://doi.org/10.3389/fnbot.2017.00002
- Adaszewski S, et al : How early can we predict Alzheimer’s disease using computational anatomy ? Neurobiol Aging 2013 ; 34(12) : 2815-2826.
- Christen M, et al. : Sur la compatibilité de la recherche guidée par les grandes données et le consentement éclairé : l’exemple du projet sur le cerveau humain. In : Mittelstadt B., Floridi L. (eds). L’éthique des données biomédicales volumineuses. Law, Governance and Technology Series 2016 ; 29. Springer, Cham
Littérature complémentaire :
- Amunts K, et al. : The Human Brain Project : Creating a European Research Infrastructure to Decode the Human Brain : Neuron 2016 ; 92 (3) : 574-581, https://doi.org/10.1016/j.neuron.2016.10.046
- Amunts K, et al. BigBrain : Un modèle de cerveau humain 3D à très haute résolution. Science 2013 ; 340 : 1472-1475, DOI : 10.1126/science.1235381, http://science.sciencemag.org/content/340/6139/1472
- Deco G, Kringelbach ML. Great Expectations : Using Whole-Brain Computational Connectomics for Understanding Neuropsychiatric Disorders. Neuron 2014 ; 84 (5) : 892-905. https://doi.org/10.1016/j.neuron.2014.08.034
InFo NEUROLOGIE & PSYCHIATRIE 2017 ; 15(6) : 46-48