Un résumé de la situation actuelle de la pharmacogénétique en psychiatrie – avec des conseils pratiques pour son utilisation, étant donné qu’une révision de la liste des analyses est entrée en vigueur depuis début 2017. Sous certaines conditions, celle-ci permet le remboursement des tests pharmacogénétiques.
La pharmacogénétique en psychiatrie s’apprête à fêter son 50e anniversaire. Dès 1969, des auteurs scandinaves avaient constaté qu’après un traitement par nortriptyline, les taux plasmatiques de cet antidépresseur tricyclique noradrénergique étaient très similaires au sein de paires de jumeaux identiques mais non dizygotes, ce qui les avait amenés à conclure que la variabilité des taux plasmatiques avait une origine génétique [1]. Parallèlement, ce travail indiquait déjà l’apparition d’interactions pharmacocinétiques qui doivent être qualifiées de facteurs environnementaux, dans la mesure où la similitude des concentrations de nortriptyline au sein des paires n’a pas été observée chez les vrais jumeaux traités par des médicaments supplémentaires.

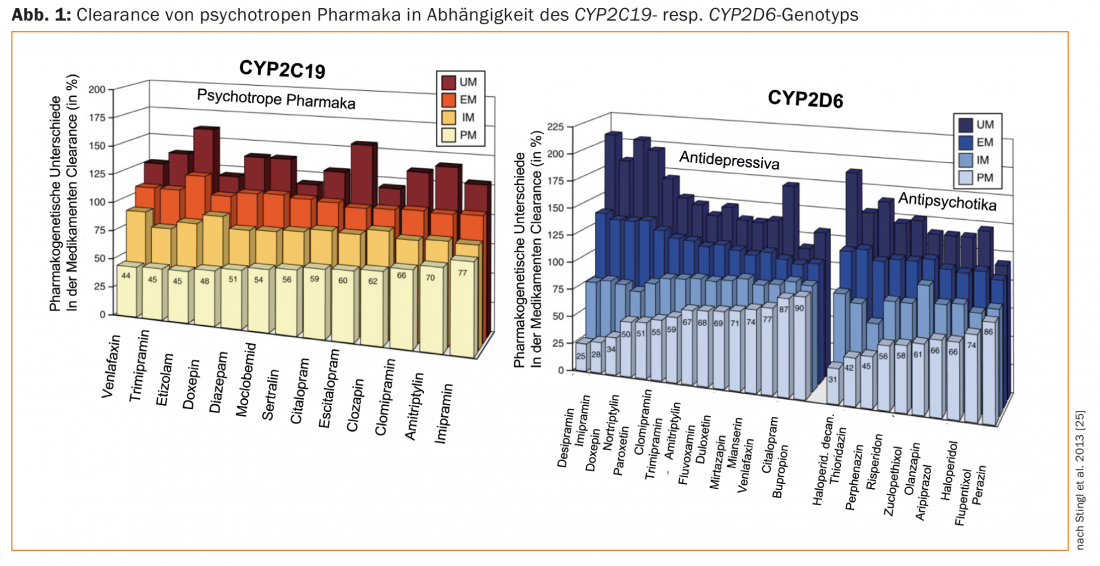
L’étape importante suivante, en 1977, a été la description de différents phénotypes, à savoir les métaboliseurs “efficaces” (EM) ou “normaux” et les “non-métaboliseurs” (n.m., aujourd’hui appelés “poor metabolisers”, PM) de l’antihypertenseur débrisoquine [2]. Une déficience génétique de la débrisoquine hydroxylase, connue aujourd’hui sous le nom d’enzyme génétiquement polymorphe cytochrome P-450 2D6, ou CYP2D6, a été suspectée chez les PM sur la base de données pharmacocinétiques. En 1988, les techniques modernes d’analyse de l’ADN ont permis de mettre en évidence des génotypes, à savoir la première détection d’un polymorphisme génétique par biologie moléculaire, en prenant l’exemple du CYP2D6 [3]. En 1981, des taux plasmatiques exceptionnellement élevés de nortriptyline ont été mesurés chez une patiente traitée par nortriptyline, dont le test pharmacogénétique de debrisoquin a révélé qu’elle était PM. De plus, malgré la dose habituelle (75 mg/jour), la patiente a souffert d’effets secondaires importants. Une diminution de la dose à 25 mg/jour a entraîné une amélioration clinique et une disparition des effets secondaires [4]. Cette étude de cas historique est considérée comme la première description psychopharmacogénétique cliniquement pertinente. Depuis, de nombreuses études ont étayé l’hypothèse selon laquelle, en plus des facteurs environnementaux et des facteurs liés à la personne du patient, des facteurs génétiques sont responsables des différences interindividuelles dans la pharmacocinétique et la pharmacodynamie des médicaments psychotropes (Fig. 1) [5]. En particulier, de nombreux variants génétiques ont été identifiés dans les enzymes impliquées dans le métabolisme de ces médicaments et leur influence sur l’activité enzymatique a été caractérisée [6,7]. Grâce à la collaboration entre le monde académique et l’industrie dans le domaine de la pharmacogénomique-pharmacogénétique, il existe déjà des biomarqueurs génétiques adaptés à une mise en œuvre de la thérapie personnalisée dans la pratique [7,8].
Cet article résume la situation actuelle de la pharmacogénétique en psychiatrie. Il contient des conseils pratiques pour leur utilisation, car la pharmacogénomique a récemment connu une innovation importante en Suisse : Le 1er janvier 2017, une révision de la liste des analyses est entrée en vigueur, permettant sous certaines conditions le remboursement des tests pharmacogénétiques par l’assurance de base.
Importance générale de la pharmacogénomique-pharmacogénétique
Sous le nom de pharmacogénomique s’est développée une discipline qui étudie l’impact des différences génétiques interindividuelles sur la pharmacocinétique, les effets thérapeutiques ainsi que les effets secondaires des médicaments [9]. Souvent, la pharmacogénomique et la pharmacogénétique sont utilisées comme synonymes. Les biomarqueurs pharmacogénétiques permettent d’estimer la réponse thérapeutique à un médicament ou sa toxicité chez un patient individuel. Il s’agit souvent de variantes génétiques d’enzymes du métabolisme des médicaments ou de protéines de transport, de récepteurs responsables de l’action des médicaments, mais aussi du complexe majeur d’histocompatibilité (antigènes leucocytaires humains, HLA ; voir l’exemple suivant de la carbamazépine).
Dans la pratique clinique, les rôles suivants sont attribués à une étude pharmacogénétique [10]:
- Éviter les surdosages qui entraînent des effets indésirables des médicaments
- Éviter les sous-dosages qui entraînent un effet thérapeutique insuffisant
- Éviter l’utilisation de médicaments chez les patients hypersensibles et exposés aux risques
- Amélioration du diagnostic différentiel, par exemple pour établir la causalité d’un effet indésirable d’un médicament.

L’objectif général est d’utiliser les connaissances issues de la pharmacogénomique-pharmacogénétique pour adapter les thérapies médicamenteuses au profil génétique d’un patient individuel. D’autres auteurs parlent à cet égard de “pharmacothérapie stratifiée” [11]. Dans ce contexte, le groupe suisse “Pharmacogénomique et thérapie personnalisée” a été récemment créé en tant que section de la Société suisse de pharmacologie clinique et de toxicologie (SSPC) afin de contribuer de manière significative à leur mise en œuvre, mais aussi de promouvoir la recherche. (encadré “Swiss Group of Pharmacogenomics and Personalised Therapy”). Il faut préciser qu’en pharmacogénétique, on n’étudie généralement pas les gènes responsables de maladies [12]. La pharmacogénomique étudie les variants génétiques qui influencent les domaines de la pharmacocinétique et de la pharmacodynamie d’un médicament. Les cibles des médicaments sont par exemple les récepteurs ou les protéines de transport des neurotransmetteurs. En fonction de la variante génétique présente, les médicaments interagissent différemment avec eux d’un point de vue pharmacologique. Les médicaments peuvent donc avoir une pharmacodynamie interindividuelle en raison des différents profils génétiques des patients traités. À cet égard, il existe de nombreuses études sur l’importance des variantes génétiques des protéines de transport des neurotransmetteurs (par exemple, le transporteur 5-HT) et des protéines de récepteur (par exemple, le récepteur de la dopamine) pour l’effet clinique des médicaments psychotropes [13,14]. Cependant, le génotypage dans ce domaine n’est actuellement pas recommandé dans la pratique quotidienne en raison de l’insuffisance des preuves [15].
La situation est différente dans le domaine de la pharmacocinétique-pharmacogénomique, où une timide mise en œuvre est déjà en cours. De nombreux auteurs et comités recommandent le génotypage des enzymes métabolisant les médicaments et, plus récemment, des protéines de transport des médicaments dans la pratique clinique quotidienne. Les quatre étapes du devenir d’un médicament dans l’organisme sont l’absorption (“absorption”), la distribution (“distribution”), le métabolisme et l’élimination (“ADME”). Auparavant, on pensait que les médicaments se diffusaient dans l’organisme par simple diffusion. Cependant, des protéines de transport ont été caractérisées entre-temps, qui accélèrent le passage des substances actives à travers les barrières membranaires et cellulaires, par exemple de l’intestin vers le sang et à travers la barrière hémato-encéphalique. L’absorption dans le foie, où se déroule une grande partie du métabolisme des médicaments, est également médiée par des protéines de transport. La glycoprotéine P (PgP) en fait partie. En tant que transporteur d’efflux, elle veille à ce que les médicaments et autres xénobiotiques, dans la mesure où ils sont des substrats de la PgP, soient rapidement éliminés du SNC et ne puissent donc pas atteindre des concentrations intracérébrales significatives. Le gène ABCB1, qui code pour la protéine PgP, présente de nombreuses variantes génétiques. En raison de ce polymorphisme génétique, il existe des individus chez lesquels la fonction de la PgP en tant que protéine de transport de certains substrats est limitée ou inexistante, ce qui affecte leur pharmacocinétique et leurs effets cliniques [16].
Comme nous l’avons mentionné au début, le système enzymatique du cytochrome P-450 joue également un rôle important dans le métabolisme des produits pharmaceutiques. Plusieurs formes telles que CYP2B6, CYP2C9, CYP2C19 et CYP2D6 présentent un polymorphisme génétique. On distingue grossièrement les PM avec aucune copie de gène active, les EM avec deux copies de gène actives – ils sont aujourd’hui appelés “métaboliseurs normaux” [17] – les métaboliseurs intermédiaires (IM) avec, par exemple, une seule copie de gène active et les métaboliseurs ultra-rapides (UM) avec plus de deux copies de gène actives comme conséquence d’une multiplication de gènes. Selon le statut métabolique génétiquement déterminé, on mesure des taux plasmatiques de médicaments extrêmement élevés (chez PM), “normaux” (chez EM, IM) ou extrêmement bas (chez UM). Il existe donc un risque accru d’effets indésirables (en cas de PM) ou de non-réponse (en cas d’UM) à la médication aux doses habituelles [18]. Pour les médicaments qui sont convertis d’une forme inactive en principe actif par une enzyme présentant un polymorphisme génétique, les groupes de risque sont inversés (non-réponse pour PM ; risque d’effets secondaires pour UM). Par exemple, la codéine, substrat du CYP2D6, est un précurseur (un “promédicament”) de la morphine : chez les UM, le risque d’intoxication à la morphine est accru après l’administration de la codéine [19].
Sources d’information pour les recommandations et les lignes directrices
Le médecin traitant fonde notamment sa stratégie thérapeutique sur les informations accessibles dans le “ résumé des caractéristiques du produit” (RCP). Celle-ci contient des informations sur le métabolisme, la pharmacocinétique et les mécanismes d’action des médicaments. Pour plusieurs médicaments, il existe désormais des indications sur l’influence des variantes pharmacogénétiques. Malgré les recommandations de la littérature scientifique, il est extrêmement rare que les informations sur les médicaments recommandent explicitement la réalisation de tests pharmacogénétiques. Les paires médicament/gène carbamazépine/HLA-A*31:01 et carbamazépine/HLA-B*15:02 constituent une exception (dans le spectre pharmacodynamique). Chez les porteurs des allèles HLA-A*31:01 ou HLA-B*15:02, le risque d’effets secondaires dermatologiques graves est nettement plus élevé que chez les non-porteurs. Il convient donc de procéder à un génotypage chez les patients chez qui un traitement par carbamazépine est prévu, notamment pour réduire le risque de survenue d’un syndrome de Stevens-Johnson. Cependant, la fréquence de ces variants de risque varie selon les groupes ethniques, c’est pourquoi, par exemple, pour les individus d’origine européenne, seul le génotypage HLA-A*31:01 est recommandé, comme décrit en détail dans la section “Carbamazépine” [20]. En revanche, la variante HLA-B*15:02 n’est fréquente que dans certaines populations asiatiques, et le génotypage n’est recommandé que chez les patients d’origine asiatique.
Une autre exception concerne un génotypage pharmacocinétique-pharmacogénétique, à savoir le couple CYP2D6/l’atomoxétine, un médicament prescrit pour le traitement du TDAH (“trouble déficitaire de l’attention avec hyperactivité”), qui est un substrat de l’enzyme. L'”Information sur le médicament” précise : “Environ 7% de tous les Caucasiens possèdent un génotype correspondant à une enzyme CYP2D6 défectueuse (appelée CYP2D6 “pauvre métaboliseur”). Les patients présentant ce génotype ont une exposition à l’atomoxétine plusieurs fois supérieure à celle des patients dont l’enzyme est fonctionnelle. Les “poor metaboliser” présentent donc un risque accru d’effets indésirables. Chez les patients dont le génotype “poor metaboliser” est connu, une dose initiale plus faible et une titration plus lente doivent être envisagées”. Malgré ces recommandations d’action, le génotypage évident avant le début du traitement n’est pas explicitement recommandé dans l’information sur le médicament. Pour le même médicament, les données pharmacocinétiques permettent par ailleurs de supposer que le risque de non-réponse à l’atomoxétine est élevé chez les patients UM (CYP2D6). [21].
Pour d’autres médicaments tels que l’aripiprazole, bien que l’information sur le médicament mentionne que cet antipsychotique est métabolisé par le CYP2D6 et que sa biotransformation est donc fortement réduite chez les PM, il n’existe aucune information ou recommandation d’action sur l’éventuel bénéfice d’un génotypage. Cependant, plusieurs groupes d’auteurs et organisations ont publié des lignes directrices (“guidelines”) avec des recommandations qui aident le médecin traitant à interpréter les résultats des tests pharmacogénétiques et à prendre des décisions sur la poursuite du traitement en tenant compte des informations pharmacogénétiques.
Des organisations telles que le Clinical Pharmacogenetics Implementation Consortium sont très utiles à cet égard. (CPIC) et la PharmGKB hébergée par l’université de Stanford (The Pharmacogenomics Knowledgebase). Ils rassemblent et analysent les connaissances scientifiques dans ce domaine, publient des recommandations sur le choix des médicaments et l’adaptation des doses de médicaments en fonction des résultats des tests pharmacogénétiques et des informations sur le lien entre certains gènes et variantes de gènes et l’effet clinique sur le résultat d’un traitement médicamenteux. PharmGKB propose également des outils d’interprétation en ligne où, après avoir saisi le génotype d’un patient, les recommandations pertinentes pour ce génotype s’affichent directement.
Il en ressort que ce n’est pas simplement pour un groupe de médicaments ayant des structures chimiques similaires ou la même indication, mais pour chaque médicament individuel qu’il faut déterminer, sur la base de données expérimentales et d’études cliniques, la pertinence de certaines variantes génétiques et l’utilité même d’un génotypage. Il faut donc définir des paires médicament/gène pour lesquelles un génotypage selon des critères précis fournit des informations pertinentes pour le traitement et orientant la clinique.
Niveaux de preuve
La PharmGKB a défini différents niveaux de preuves qui reflètent la force des données scientifiques soutenant un lien entre les variantes génétiques et un résultat de traitement particulier (effet, effets secondaires) pour des paires gène/médicament individuelles (voir encadré).

Les études pharmacogénétiques sont difficiles à réaliser car, en fonction de la fréquence des allèles étudiés dans une population ou de l’effet secondaire étudié, il faut inclure un très grand nombre de patients dans l’étude pour obtenir des résultats statistiquement significatifs. La PharmGKB a donc défini quatre niveaux d’évidence différents [22,23]. Chaque annotation clinique (“annotation”) contient donc une information sur le niveau atteint pour le couple médicament/gène concerné.
Le tableau 1 présente une compilation des principaux médicaments utilisés en psychiatrie, leurs protéines impliquées dans le métabolisme et le transport, ainsi que les niveaux de preuve clinique des associations. Idéalement, les niveaux de preuve devraient également se refléter dans les recommandations figurant dans les informations sur les médicaments. En outre, la Food and Drug Administration (FDA) des États-Unis a publié une liste de 200 médicaments dont l’information sur le médicament fournit des informations sur les biomarqueurs pharmacogénomiques.
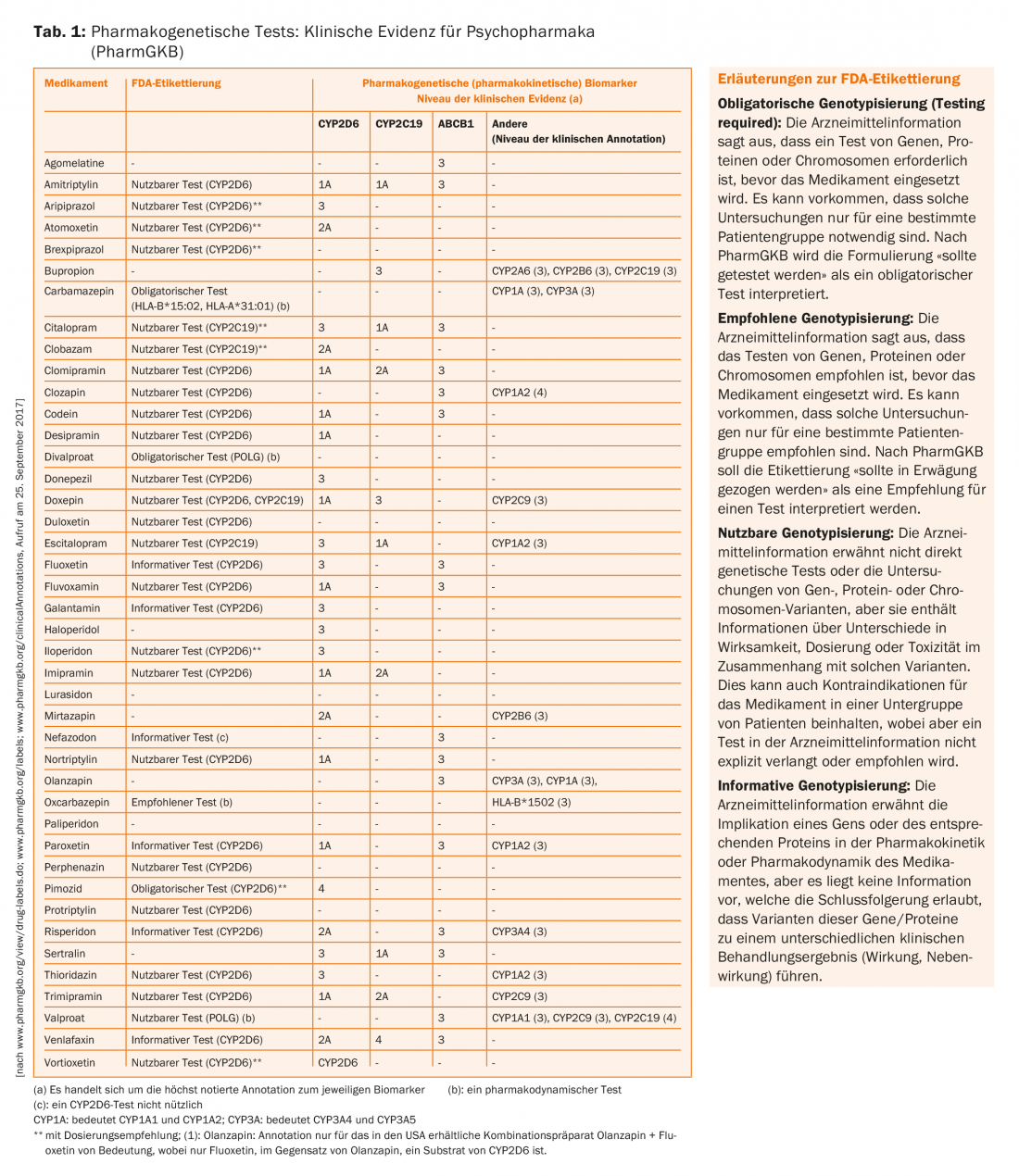
Avec 26 biomarqueurs, la psychiatrie occupe la deuxième place après l’oncologie en termes de fréquence (tableau 1) [24]. Il faut souligner qu’en oncologie, on étudie très souvent la génétique de la tumeur pour estimer l’effet d’un médicament (par exemple, certaines mutations du récepteur de l’EGF doivent être présentes dans les cellules tumorales pour que le gefitinib puisse agir), mais celle-ci diffère généralement de la génétique du patient. En revanche, les questions de pharmacogénétique concernant le métabolisme, le transport et l’action des médicaments non oncologiques utilisent les informations génétiques des cellules du corps du patient.
Le tableau 1 présente également des annotations d’informations sur les médicaments de la FDA pour les psychotropes, telles qu’elles ont été résumées par PharmGKB. Des annotations équivalentes, mais moins complètes et non présentées ici, sont également disponibles auprès de l’Agence européenne des médicaments (EMA), de l’Agence japonaise des produits pharmaceutiques et des dispositifs médicaux (PMDA), de Santé Canada Santé Canada (HCSC) et d’autres agences. De cette compilation, on peut résumer ce qui suit :
- Pour les paires de médicaments/gènes telles que carbamazépine/HLA-A*31:01 allèle et carbamazépine/HLA-B*15:02 allèle, mais aussi pour certains antidépresseurs (par ex. amitriptyline)/CYP2D6 et/ou CYP2C19, il existe des recommandations de haut niveau (1A)
- Les paires de médicaments/gènes liées aux protéines de transport telles que ABCB1 (PgP) (par exemple, citalopram/ABCB1) n’atteignent que le niveau de preuve 3.
- Pour pratiquement tous les couples médicament/gène, les tests pharmacogénétiques sont indiqués comme étant “actionnables”, c’est-à-dire utilisables ou orientés, mais ils ne sont pas nécessairement prescrits ou considérés comme obligatoires. Ce n’est que pour la carbamazépine, le divalproate (mais curieusement pas pour le valproate – cela pourrait être la conséquence d’un manque de données issues d’essais cliniques pour atteindre ce niveau de recommandation) et l’antipsychotique pimozide, qui n’est plus autorisé en Suisse, que la FDA qualifie de “nécessaire” (“required”) un test pharmacogénétique.
Recommandations pour les tests pharmacogénétiques en psychiatrie
Les revues sur la pharmacogénétique des médicaments psychotropes et les lignes directrices sur les tests pharmacogénétiques en psychiatrie portent pour la plupart sur le génotypage du CYP2D6 et du CYP2C19chez les patients traités par des antidépresseurs et des antipsychotiques [25–29]. Les médicaments se distinguent par leurs propriétés en tant que substrats de ces enzymes. Cela explique les différences importantes dans les recommandations de dose entre les différents médicaments, compte tenu des génotypes des patients (UM, EM, IM, PM ; Fig. 1 ; [25]). Par exemple, pour la paroxétine, un substrat du CYP2D presque exclusif, la dose doit être réduite à seulement environ 50% de la dose habituelle pour un PM (CYP2D6), et augmentée de plus de 50% pour un UM. En revanche, dans le cas du citalopram, cette enzyme joue un rôle mineur et il n’est donc pas nécessaire d’adapter la dose en fonction du génotype CYP2D6. En revanche, le CYP2C19 joue un rôle majeur dans le métabolisme de cet antidépresseur – selon le génotype du CYP2C19, la dose doit être augmentée (UM) ou diminuée (PM) (Fig. 1).
Une attention particulière doit être accordée à la recommandation d’auteurs suisses de renom qui, au nom de la Société suisse pour l’anxiété et la dépression (SSAD), de la Société suisse de psychiatrie biologique (SSPB) et de la Société suisse de psychiatrie et de psychothérapie (SSPP), préconisent le génotypage des ABCB1 recommander chez tous les patients dépressifs à qui l’on a prescrit un traitement par antidépresseurs, mais dont l’effet thérapeutique est insuffisant [30]. Cette recommandation a une importance en termes de politique de santé et d’économie, car elle pourrait avoir pour conséquence que des milliers de patients dépressifs soient génotypés chaque année, qu’ils soient ou non traités par un substrat PgP.
Certains antidépresseurs sont des substrats de la PgP (citalopram), mais d’autres ne le sont pas (mirtazapine). Chez les patients dépressifs traités par antidépresseurs, une étude rétrospective a montré que le polymorphisme génétique du gène ABCB1était associé à la réponse au traitement chez ceux qui étaient traités par les substrats PgP amitriptyline, paroxétine, venlafaxine ou citalopram [31]. La distribution des génotypes de la variante ABCB1(Single Nucleotide Polymorphism, SNP) rs2032583 était particulièrement importante. Il y avait beaucoup moins de patients non réformés parmi les porteurs de l’allèle C de cette variante (25%) que parmi les patients qui n’étaient pas porteurs de l’allèle C (62%). Des résultats similaires ont été observés dans les calculs liés au SNP rs2235015. Parmi les porteurs de l’allèle T de cette variante, il y avait beaucoup moins de non-remplaceurs par rapport aux non-porteurs (“non-carriers”). De telles associations n’ont pas été observées dans un groupe de comparaison de patients non traités avec des substrats PgP.
Il convient de préciser qu’un seul antidépresseur a été prescrit pour ce groupe, à savoir la mirtazapine. Dans une méta-analyse, la même équipe a conclu que les SNPs rs2032583 et rs2235015 étaient significativement associés à l’effet clinique [32], alors que l’analyse d’autres SNPs ne montrait pas une telle association. Mais en même temps, dans une autre étude, les chercheurs ont conclu que le maintien de taux plasmatiques thérapeutiques d’antidépresseurs est une mesure nécessaire en plus du génotypage ABCB1 [33].
Certains auteurs de la méta-analyse susmentionnée ont récemment publié une autre analyse, beaucoup plus complète, de 32 études sur le lien entre le polymorphisme ABCB1et l’effet clinique (effet thérapeutique, tolérance) des antidépresseurs. [32]Les associations les plus élevées sont obtenues lorsque rs2032583 et rs2235040 sont analysés, mais elles sont déjà plus faibles lorsque rs2032582 est pris en compte, et encore plus faibles lorsque d’autres SNP sont pris en compte, comme le rs2235015 mentionné ci-dessus. Dans l’ensemble, les auteurs de l’analyse ont conclu que les études menées jusqu’à présent n’étaient pas suffisantes pour démontrer un bénéfice clinique du génotypage ABCB1dans le traitement médicamenteux de la dépression [34].
D’autres études cliniques sont demandées, mais aussi celles qui démontrent une importance fonctionnelle de ces variants ABCB1qui se trouvent en dehors de la région du gène codant pour la protéine. Cela pourrait être étudié à l’aide de techniques d’imagerie afin de déterminer si les patients présentant différents génotypes ABCB1 ou combinaisons de variants (haplotypes) ont une absorption différente des médicaments dans le cerveau après l’administration d’antidépresseurs. Une analyse indépendante de la littérature confirme qu’à l’heure actuelle, la généralisation du génotypage ABCB1en pratique clinique quotidienne est prématurée [16]. C’est également la conclusion à laquelle sont parvenus récemment des membres de la Commission des médicaments du corps médical allemand [35].
Pour le médecin praticien, cette controverse est déroutante, d’autant plus qu’un groupe d’auteurs doute, sur la base de leurs recherches, que l’amitriptyline soit un substrat de la Pgp chez l’homme [36]. Il n’est pas non plus facile de savoir quels variants génétiques sont étudiés par les laboratoires spécialisés qui proposent des analyses pharmacogénétiques d’ABCB1 . Un génotypage ABCB1 de routine d’une majorité de patients dépressifs traités par médicaments n’est donc clairement pas recommandé à l’heure actuelle, d’autant plus que des études sur la sensibilité et la spécificité du test font également défaut.
Importance du règlement du 1er janvier 2017 pour les tests pharmacogénétiques
Le 1er Le 1er janvier 2017, une ordonnance révisée est entrée en vigueur en Suisse, selon laquelle les tests pharmacogénétiques sont pris en charge par l’assurance obligatoire des soins si certaines conditions sont remplies (ordonnance du DFI du 29. septembre 1995 sur les prestations dans l’assurance obligatoire des soins en cas de maladie (OPAS, état au 1er janvier 2017). D’un point de vue pratique, ces analyses sont désormais incluses dans la Liste fédérale des analyses (annexe 3 de l’OPAS). Le thème des tests pharmacogénétiques est traité dans la liste des analyses du 1er juillet 2017 sous les positions 2150.10, 2250.10, 2271.01, 2547.01 (vérifiée le 14.7.2017). Il convient de distinguer deux conditions :
- Un petit nombre de tests pharmacogénétiques peuvent être prescrits par tous les médecins, quel que soit leur titre de spécialiste, et remboursés par l’assurance maladie. Une liste de ces analyses en relation avec des médicaments définis (il s’agit donc à chaque fois de paires médicament/gène) est établie par la Société Suisse de Pharmacologie et Toxicologie Cliniques (SSPTC), 9.6.2016, version 1) :
- Abacavir (HLA-B*57:01) ;
- carbamazépine (HLA-A*31:01 et HLA-B*15:02) ;
- 6-mercaptopurine, azathioprine (TPMT) ;
- 5-fluorouracile, capécitabine (DPYD) ;
- Irinotécan (UGT1A1).
Cette liste est mise à jour chaque année par la SSCPT, qui s’appuie à la fois sur la littérature scientifique actuelle et sur les preuves obtenues dans le cadre de l’expérience clinique pour choisir les paires gène/médicament. Pour la psychiatrie et la neurologie, il est important que tout psychiatre, neurologue, mais aussi médecin généraliste, puisse (et doive) effectuer un génotypage HLA-A*31:01 et HLA-B*15:02 (pour les patients d’origine asiatique) lors d’un nouveau traitement prévu à la carbamazépine, et que les caisses d’assurance maladie remboursent au patient le coût du test.
2. pour les tests pharmacogénétiques destinés à être prescrits pour d’autres paires médicament/gène que celles indiquées ci-dessus, les deux limitations suivantes dans la liste des analyses sont particulièrement importantes :
- Limitation 1 : “Uniquement en cas d’indication d’administration d’un médicament ou de survenue d’un effet indésirable médicamenteux ou d’une diminution ou d’une absence d’efficacité thérapeutique lors d’un traitement par un médicament pour lequel il existe un lien scientifiquement prouvé entre des effets indésirables médicamenteux significatifs (y compris des effets toxiques) ou une diminution ou une absence d’efficacité thérapeutique et les mutations génétiques étudiées”.
- Limitation 4 : “Pour les médicaments qui ne figurent pas sur la liste de la SSCPT, prescription de l’analyse uniquement par des médecins titulaires d’un titre postgrade fédéral en pharmacologie et toxicologie cliniques conformément à la loi fédérale du 23 juin 2006 sur les professions médicales universitaires (loi sur les professions médicales, LPMéd ; RS 811.11)”.
D’un point de vue éthique, il convient également de noter la limite 3, selon laquelle les tests ne doivent pas servir à établir un diagnostic ou à rechercher une prédisposition à une maladie génétique.
Conseils pratiques
Cet article montre que la pharmacogénomique en psychiatrie aura une importance croissante pour la planification et la mise en œuvre de la pharmacothérapie. Grâce à la révision de la liste des analyses, les analyses pharmacogénétiques pertinentes en psychiatrie sont disponibles à la charge de l’assurance maladie obligatoire lorsqu’un spécialiste en pharmacologie clinique et toxicologie a déterminé la nécessité du test. Pour planifier des tests pharmacogénétiques et interpréter leurs résultats de manière optimale, il est nécessaire de disposer de connaissances appropriées. Ce sujet devrait donc être davantage pris en compte dans la formation des spécialistes en psychiatrie et en psychothérapie, ainsi que dans les programmes de formation continue et de perfectionnement [37–39]. Des cours destinés au grand public sont également nécessaires pour promouvoir la compréhension générale de la pharmacogénomique et optimiser sa mise en œuvre [40]. Il est recommandé de consulter des ouvrages spécialisés qui non seulement résument les bases génétiques, mais qui contiennent également des conseils sur l’approche pratique et des exemples cliniques [11]. En outre, compte tenu de la difficulté de la matière et en conséquence du nouveau règlement, une collaboration avec des pharmacologues et toxicologues cliniques est particulièrement précieuse. Il est également recommandé de collaborer avec des spécialistes de laboratoire formés en génétique pour l’exécution pratique.
Les indications d’un test pharmacogénétique sont par exemple : Des taux plasmatiques de médicaments plus élevés ou plus faibles que ceux normalement attendus à la dose administrée, suggérant que le patient présente une particularité génétique dans son métabolisme. Le médicament doit donc être un substrat de l’enzyme cytochrome P-450 génétiquement polymorphe à tester. Lorsqu’un patient est co-médiqué avec des inhibiteurs, il faut également tenir compte du fait que des taux plasmatiques élevés peuvent simuler une déficience génétique du métabolisme en raison d’une interaction médicamenteuse. De même, la co-médication avec un médicament inducteur d’enzymes peut entraîner une baisse des taux plasmatiques qui n’est pas d’origine génétique. Les tests pharmacogénétiques doivent donc généralement être considérés comme un complément au suivi thérapeutique (TDM). (Fig. 2). Cette approche est également recommandée par l’AGNP (Arbeitsgemeinschaft für Neuropsychopharmakologie und Pharmakopsychiatrie), qui est en train de rééditer un “Consensus Guideline” pour le TDM : il décrit l’utilisation optimale du TDM seul, mais aussi en combinaison avec des tests pharmacogénétiques. [41,42].
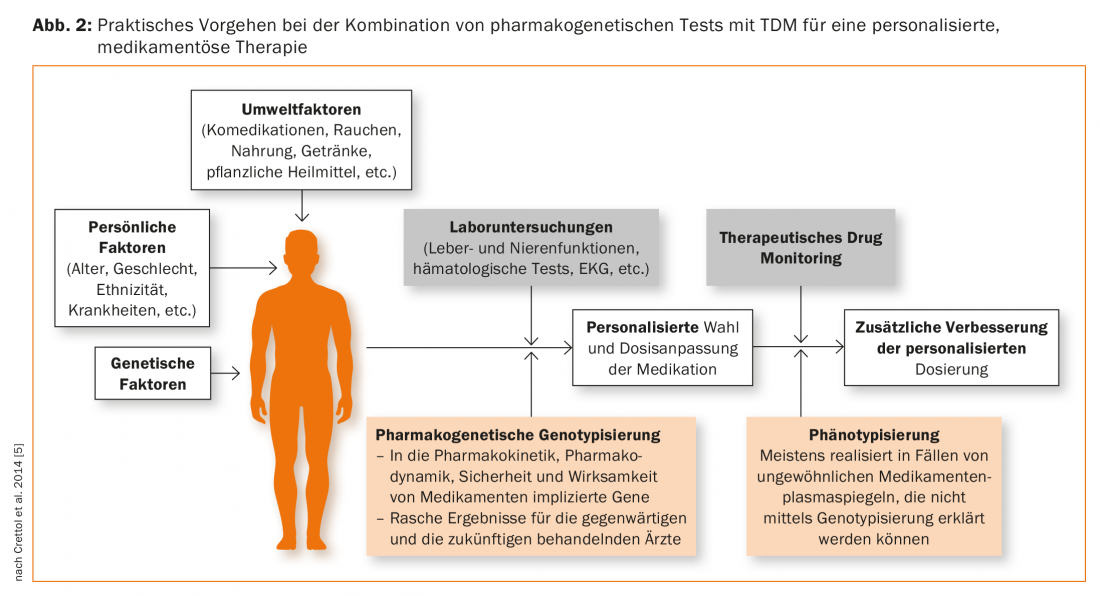
Le génotypage donne des résultats qui restent valables toute la vie et constitue donc une sorte de “marqueur de trait”. En revanche, le résultat d’un phénotypage, comme la détermination d’un taux métabolique, doit être considéré comme un “marqueur d’état”. Pour ce faire, on administre au patient une substance qui est un substrat de l’enzyme à tester, puis on recueille du sang ou de l’urine, selon le protocole, et on détermine généralement la substance mère et le métabolite. Le rapport métabolite/substance mère fournit un quotient métabolique, qui est une mesure de la capacité métabolique de l’enzyme testée, comme illustré ci-dessus par l’exemple du test de débris. Le résultat peut fournir des informations sur le profil génétique du patient, dans la mesure où EM et PM se distinguent par leur quotient métabolique. Cependant, si le patient est sous l’influence de médicaments ou d’autres substances étrangères qui inhibent ou induisent l’enzyme, cette influence environnementale masque la composante génétique du métabolisme de la substance testée. En psychiatrie, les phénotypages suivants sont courants : CYP2D6 (dextrométhorphane comme substance test), CYP2C19 (oméprazole), CYP1A2 (caféine), CYP2B6 (bupropion), CYP3A (midazolam) [43,44]. Le phénotypage est particulièrement utile pour les enzymes telles que le CYP1A2 et le CYP3A (CYP3A4), dont l’activité varie considérablement d’un individu à l’autre, mais pour lesquelles aucun polymorphisme génétique cliniquement significatif n’est connu et pour lesquelles le génotypage n’est donc pas utile. La fexofénadine [44] ou la digoxine [45] ont été proposées pour le phénotypage de l’activité ABCB1. Selon la question posée, plusieurs échantillons de test pour les différentes enzymes ou transporteurs peuvent être administrés simultanément sous forme de “cocktail” [44,45]. Le phénotypage est donc un complément au TDM et au génotypage (Fig. 2) [46], car le résultat fournit une information sur l’état actuel de l’activité de l’enzyme ou du transporteur au moment du test [42].
Les informations telles que les taux plasmatiques des médicaments, les comédications, les comorbidités, le type d’effets secondaires éventuels, les renseignements sur la réponse thérapeutique au médicament, sont utiles et nécessaires au pharmacologue clinique pour justifier une prescription. Pour les questions de pharmacogénétique, les centres suisses de pharmacologie clinique et de toxicologie proposent des consultations et les patients peuvent être envoyés en consultation pour poser l’indication d’une analyse pharmacogénétique.
Conclusions
Le temps est maintenant venu d’avancer dans l’implémentation des tests pharmacogénétiques dans la psychopharmacothérapie. Ils peuvent constituer un outil précieux en complément des essais cliniques ainsi que du TDM (Fig. 2). Les autorités telles que la FDA et l’EMA, ainsi que des organismes scientifiques tels que PharmGKB et CPIC, publient régulièrement des recommandations pour l’utilisation clinique des tests pharmacogénétiques. Il existe donc désormais des lignes directrices développées par des organismes indépendants et adaptées à une utilisation clinique quotidienne. Ils favorisent la mise en œuvre appropriée et l’interprétation des résultats des tests pharmacogénétiques et donc une thérapie optimisée en fonction du génotype [48]. Il est important de replacer le résultat génétique dans le contexte global du traitement du patient, c’est-à-dire de tenir compte des influences non génétiques telles que la comédication lors de l’adaptation du traitement. Des lignes directrices adaptées à chaque région sont également nécessaires pour permettre une application économique et clinique adaptée aux conditions locales, suite à la décision politique de rendre les tests pharmacogénétiques remboursables sous certaines conditions et donc plus accessibles. Une mauvaise utilisation peut conduire à des résultats décevants et, à moyen terme, le médecin praticien perdra confiance dans la pharmacogénomique et la thérapie personnalisée [49]. Comme mentionné ci-dessus, le thème de la pharmacogénomique en psychiatrie devrait être davantage pris en compte dans la formation initiale et continue des médecins spécialistes en psychiatrie et psychothérapie [37–39]. Il convient donc d’encourager la collaboration entre les psychiatres et les médecins praticiens d’une part, et le spécialiste en pharmacologie clinique d’autre part. Parallèlement, de nombreuses questions restent en suspens, ce qui explique pourquoi les études cliniques en pharmacogénétique, notamment, doivent être planifiées et réalisées de manière adéquate.
Littérature :
- Alexanderson B, Evans DA, Sjöqvist F : Taux plasmatiques de nortriptyline à l’état d’équilibre chez les jumeaux : influence des facteurs génétiques et du traitement médicamenteux. British Medical Journal 1969 ; 4(686) : 764-768.
- Mahgoub A, et al : Polymorphic hydroxylation of debrisoquine in man. Lancet 1977 : 584-6.
- Gonzalez FJ, et al : Caractérisation du défaut génétique commun chez les humains déficients en métabolisme de la debrisoquine. Nature 1988 ; 331 : 442-446.
- Bertilsson L, et al : Slow hydroxylation of nortriptyline and concomiting poor debrisoquine hydroxylation : clinical implications. Lancet 1981 ; i : 560-561.
- Crettol S, et al : Pharmacogenomics in psychiatry : from therapeutic drug monitoring to genomic medicine. Clin Pharmacol Ther 2014 ; 95(3) : 254-257.
- Pratt V, et al : Medical Genetics Summaries. Bethesda (Md), USA : National Center for Biotechnology Information ; 2017. 416 p.
- Zhang G, Nebert DW : Médecine personnalisée : prédiction du risque génétique de la réponse aux médicaments. Pharmacol Ther 2017 ; 175 : 75-90.
- Relling MV, Evans WE : Pharmacogénomique en clinique. Nature 2015 ; 526(7573) : 343-350.
- Patil J : Pharmacogenetics and Pharmacogenomics : A Brief Introduction. Journal of Pharmacovigilance 2015 ; 3 : e139.
- Sim SC, Ingelman-Sundberg M : Biomarqueurs pharmacogénomiques : nouveaux outils dans la thérapie médicamenteuse actuelle et future. Trends Pharmacol Sci 2011 ; 32(2) : 72-81.
- Dingermann T, Zündorf I : Pharmacothérapie stratifiée. Bases génétiques, approche pratique. Eschborn, Allemagne : Govi ; 2017. 339 p.
- Gandal MJ, et al : The road to precision psychiatry : translating genetics into disease mechanisms. Nat Neurosci 2016 ; 19(11) : 1397-1407.
- Lohoff FW, Ferraro TN : Considérations pharmacogénétiques dans le traitement des troubles psychiatriques. Expert Opin Pharmacother 2010 ; 11(3) : 423-439.
- Budde M, et al : Pharmacogenomic aspects of bipolar disorder : An update. Eur Neuropsychopharmacol 2017 ; 27(6) : 599-609.
- Malhotra AK, Zhang JP, Lencz T : Pharmacogenetics in psychiatry : translating research into clinical practice. Mol Psychiatry 2012 ; 17(8) : 760-769.
- Wolking S, et al. : Impact des polymorphismes génétiques de l’ABCB1 (MDR1, P-glycoprotéine) sur la disposition à prendre des médicaments et les implications cliniques potentielles : mise à jour de la littérature. Clinical Pharmacokinetics 2015 ; 54(7) : 709-735.
- Caudle KE, et al : Standardizing terms for clinical pharmacogenetic test results : consensus terms from the Clinical Pharmacogenetics Implementation Consortium (CPIC). Genet Med 2017 ; 19(2) : 215-223.
- Hicks JK, et al : Clinical pharmacogenetics implementation consortium guideline (CPIC) for CYP2D6 and CYP2C19 genotypes and dosing of tricyclic antidepressants : 2016 update. Clin Pharmacol Ther 2017 ; 102(1) : 37-44.
- Gasche Y, et al : Codeine intoxication associated with ultrarapid CYP2D6 metabolism. N Engl J Med 2004 ; 351(27) : 2827-2831.
- McCormack M, et al : HLA-A*3101 et les réactions d’hypersensibilité induites par la carbamazépine chez les Européens. N Engl J Med 2011 ; 364(12) : 1134-1143.
- de Leon J : Translating Pharmacogenetics to Clinical Practice : Do Cytochrome P450 2D6 Ultrarapid Metabolizers Need Higher Atomoxetine Doses ? J Am Acad Child Adolesc Psychiatry 2015 ; 54(7) : 532-534.
- Whirl-Carrillo M, et al : Pharmacogenomics knowledge for personalized medicine. Clin Pharmacol Ther 2012 ; 92(4) : 414-417.
- McDonagh EM, et al : De l’acquisition de connaissances pharmacogénomiques aux applications cliniques : le PharmGKB en tant que ressource de biomarqueurs pharmacogénomiques cliniques. Biomark Med 2011 ; 5(6) : 795-806.
- Dickmann LJ, Ware JA : Pharmacogénomics in the age of personalized medicine. Drug Discov Today Technol 2016 ; 21-22 : 11-6.
- Stingl JC, Brockmoller J, Viviani R : Variabilité génétique des enzymes de métabolization des médicaments : le double impact sur le traitement psychiatrique et la régulation de la fonction cérébrale. Mol Psychiatry 2013 ; 18(3) : 273-287.
- Stingl JC, Brockmoller J : [Personalised pharmacogenetics. Evidence-based guidelines and clinical application of pharmacogenetic diagnostics]. Bundesgesundheitsblatt, recherche en santé, protection de la santé. 2013 ; 56(11) : 1509-1521.
- Ravyn D, et al : CYP450 pharmacogenetic treatment strategies for antipsychotics : a review of the evidence. Schizophr Res 2013 ; 149(1-3) : 1-14.
- Fabbri C, et al : Progrès et perspectives en pharmacogénétique des médicaments antidépresseurs. Expert Opin Drug Metab Toxicol 2016 ; 12(10) : 1157-1168.
- Spina E, de Leon J : Applications cliniques du génotypage du CYP en psychiatrie. J Neural Transm (Vienne) 2015 ; 122(1) : 5-28.
- Holsboer-Trachsler E, et al. : Traitement aigu des épisodes dépressifs. Swiss Medical Forum 2016 ; 16(35) : 716-724.
- Uhr M, et al : Les polymorphismes dans le gène transporteur de médicaments ABCB1 prédisent la réponse au traitement antidépresseur dans la dépression. Neuron 2008 ; 57(2) : 203-209.
- Breitenstein B, et al : ABCB1 gene variants and antidepressant treatment outcome : A meta-analysis. Am J Med Genet B Neuropsychiatr Genet 2015 ; 168B(4) : 274-283.
- Breitenstein B, et al : Association des variants du gène ABCB1, concentration d’antidépresseurs dans le plasma, et réponse au traitement : résultats d’une étude clinique randomisée. J Psychiatr Res 2016 ; 73 : 86-95.
- Bruckl TM, Uhr M : Génotypage de l’ABCB1 dans le traitement de la dépression. Pharmacogénomics 2016 ; 17(18) : 2039-2069.
- Bschor T, et al. :[Genetic tests for controlling treatment with antidepressants]. Neurologue 2017 ; 88(5) : 495-499.
- O’Brien FE, et al : Human P-glycoprotein differentially affects antidepressant drug transport : relevance to blood-brain barrier permeability. Int J Neuropsychopharmacol 2013 ; 16(10) : 2259-2272.
- Baumann P, et al. : A proposal for a psychopharmacology-pharmacotherapy catalogue of learning objectives and a curriculum in Europe. World J Biol Psychiatry 2017 ; 18(1) : 29-38.
- Whirl-Carrillo M, et al : Novel Disease-Drug Database Demonstrating Applicability for Pharmacogenomic-Based Prescribing. Clin Pharmacol Ther 2016 ; 100(6) : 600-602.
- Collins SL, Carr DF, Pirmohamed M : Advances in the Pharmacogenomics of Adverse Drug Reactions. Drug Saf 2016 ; 39(1) : 15-27.
- Green ED, Guyer MS ; National Human Genome Research I : Charting a course for genomic medicine from base pairs to bedside. Nature 2011 ; 470(7333) : 204-213.
- Hiemke C, et al : AGNP consensus guidelines for therapeutic drug monitoring in psychiatry : Update 2011. Pharmacopsychiatry 2011 ; 44(6) : 195-235.
- Hiemke C, et al : Consensus Guidelines for Therapeutic Drug Monitoring in Neuropsychopharmacology : Update 2017. Pharmacopsychiatry 2017 Sep 14. doi : 10.1055/s-0043-116492. [Epub ahead of print]
- Hiemke C, Shams M : Phenotyping and genotyping of drug metabolism to guide pharmacotherapy in psychiatry. Curr drug deliv 2013 ; 10(1) : 46-53.
- Bosilkovska M, et al. : Geneva cocktail for cytochrome p450 and P-glycoprotein activity assessment using dried blood spots. Clin Pharmacol Ther 2014 ; 96(3) : 349-359.
- Fuhr U, Jetter A, Kirchheiner J : Procédures de phénotypage appropriées pour les enzymes de métabolization des médicaments et les transporteurs chez l’homme et leur utilisation simultanée dans l’approche “cocktail”. Clin Pharmacol Ther 2007 ; 81(2) : 270-283.
- Crettol S, et al : Pharmacogenomics in psychiatry – from TDM to genomic medicine. Clin Pharmacol Ther 2014 ; 95(3) : 254-257.
- Baumann P, et al : Epileptiform seizure following sertraline treatment in an adolescent suffering from obsessional compulsive disorder and presenting a rare pharmacogenetic status. J Clin Psychopharmacol 2006 ; 26(6) : 679-681.
- Amstutz U, Carleton BC : Test pharmacogénétique : le temps des lignes directrices pour la pratique clinique. Clin Pharmacol Ther 2011 ; 89(6) : 924-927.
- de Leon J : Pharmacogenetic Tests in Psychiatry : From Fear to Failure to Hype. J Clin Psychopharmacol 2016 ; 36(4) : 299-304.
- Mlakar V, et al : Pharmacogenomics in Pediatric Oncology : Review of Gene-Drug Associations for Clinical Use. Int J Mol Sci 2016 ; 17(9) : 1502.
InFo NEUROLOGIE & PSYCHIATRIE 2017 ; 15(6) : 21-30