Dans le domaine du diagnostic des tumeurs, le terme de biopsies liquides désigne l’analyse de l’ADN acellulaire circulant (cfDNA) et des cellules tumorales circulantes. De nos jours, il est possible de détecter les mutations d’une tumeur de manière peu invasive grâce à l’analyse du cfDNA dans les tumeurs avancées. Pour les analyses cfDNA, il convient d’utiliser du plasma et non du sérum. Les cellules tumorales circulantes pourront être utilisées dans un avenir proche pour l’analyse prédictive des marqueurs, comme par exemple pour déterminer le variant d’épissage AR-V7 dans le cancer de la prostate résistant à la castration.
Bien que le terme de “biopsies liquides” soit relativement nouveau dans le contexte du diagnostic moléculaire des tumeurs, il est largement entré dans les mœurs des pathologistes et des oncologues. Dans le diagnostic des tumeurs, ce terme a d’abord été utilisé pour l’analyse des cellules tumorales circulantes (“circulating tumor cells”, CTC) et plus tard seulement pour celle de l’ADN libre de cellules circulantes (“circulating cell free DNA”, cfDNA). En résumé, il s’agit d’un terme générique désignant le prélèvement de matériel sur une tumeur solide par simple prélèvement d’un échantillon de sang. Néanmoins, le terme “biopsie liquide” ne se limite pas exclusivement à l’utilisation dans le diagnostic des tumeurs : Dans le diagnostic prénatal, les prélèvements sanguins permettent de détecter des anomalies chromosomiques chez le fœtus.
Intérêt pour la détection peu invasive
Les cellules tumorales circulantes (CTC) et l’ADN acellulaire circulant (cfDNA) sont étudiés depuis des décennies et font l’objet de discussions dans la littérature en tant que marqueurs diagnostiques et pronostiques potentiels. La première description de la CTC a eu lieu au 19e siècle par l’Australien J. Ashworth (1869), celle de l’ADNcf un peu moins d’un siècle plus tard par les deux Français P. Mandel et P. Metais (1948).
Les CTC peuvent provenir à la fois de la tumeur primaire et des métastases. Il n’est pas encore clairement établi si la libération de CTC est un processus actif et fait donc partie d’un programme biologique d’une tumeur, ou si la libération est aléatoire. En revanche, on sait qu’une partie de ces cellules a la capacité de survivre dans la circulation sanguine et de se reproduire ou de se multiplier à un autre endroit du corps humain. de former une métastase. La question de savoir si des cellules individuelles suffisent pour cela ou si ce ne sont pas des groupes de cellules tumorales qui sont responsables du processus de métastases hématogènes à distance est controversée [1].
En revanche, l’ADN acellulaire circulant (cfDNA) provient de cellules qui meurent (apoptotiques ou nécrotiques). L’ADNcf se compose à la fois d’ADN provenant de cellules tumorales (ADN tumoral circulant sans cellules, abrégé en ADNct) et de cellules saines (ADN normal circulant sans cellules, abrégé en ADNcn). La question de savoir si l’ADNcf exerce une fonction dans le sang est controversée. Comparé aux CTC (1-2,5 heures), le cfDNA dispose d’une demi-vie relativement courte de 15-30 minutes.
Tant le CTC que le cfDNA/ctDNA gagnent fortement en importance pour le diagnostic des tumeurs grâce aux nouvelles possibilités d’analyses génomiques. Le séquençage de nouvelle génération (NGS) permet aujourd’hui de séquencer des génomes entiers dans un délai très court. Depuis l’utilisation de cette technologie dans un grand nombre d’études cliniques, le nombre de marqueurs génomiques potentiellement prédictifs a fortement augmenté et, par conséquent, l’intérêt pour une détection peu invasive de ces marqueurs prédictifs dans le sang a également augmenté.
Déterminer les mutations de manière significative
Par rapport au séquençage Sanger conventionnel, le NGS permet de séquencer de plus grandes régions simultanément et surtout avec une grande profondeur (redondance). Cette profondeur est nécessaire si l’on veut détecter des mutations sous-clonales ou si l’ADN tumoral ne représente qu’une fraction de l’ADN total à analyser. C’est le cas de l’analyse de l’ADNc : en fonction de l’entité tumorale et du stade de la maladie tumorale, la proportion d’ADNc dans le sang peut varier considérablement, voire atteindre quelques pour mille.
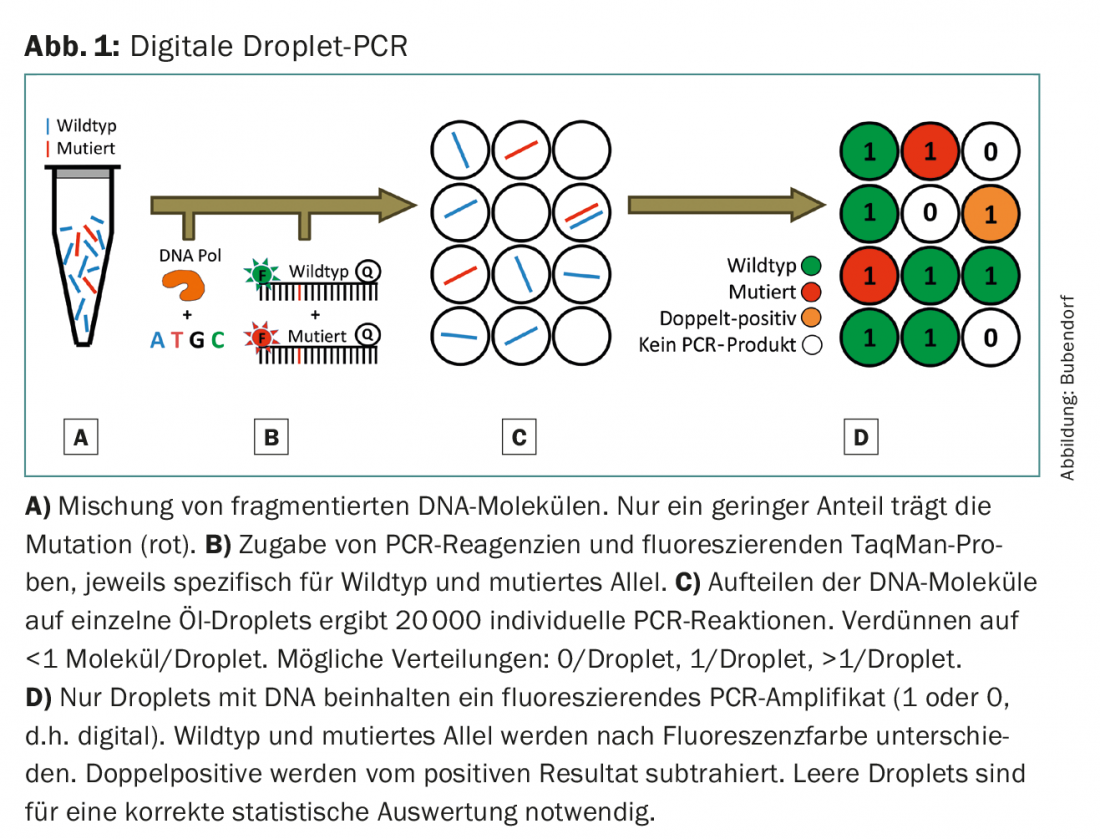
Avec la plupart des appareils de séquençage NGS, les protocoles standard permettent de détecter des mutations qui ne représentent que 2 à 5 % de l’ADN total. Cependant, l’ADNc est souvent inférieur à ce seuil, en particulier dans les tumeurs non encore avancées ou chez les patients sous traitement. Contrairement au NGS, la PCR numérique permet de déterminer une mutation dans cette zone très sensible : à l’aide de sondes spécifiques marquées à la fluorescence, certaines mutations peuvent être déterminées de manière significative dans une plage de 0,01%. La procédure et la technique sont décrites à l’aide de l’exemple de la PCR numérique en gouttelettes (fig. 1). L’inconvénient de cette méthode est qu’il faut effectuer une réaction distincte pour chaque mutation à déterminer. Il n’est donc pas recommandé d’utiliser cette méthodologie pour une étude générale du génome, mais uniquement pour l’étude de mutations individuelles.
Cellules tumorales circulantes (CTC)
Au cours de la dernière décennie, de nombreuses études ont montré que la détection d’une plus grande quantité de CTC dans le sang des patients atteints d’un cancer avancé était associée à une mauvaise survie [2]. Dans ce contexte, la FDA américaine a approuvé le relevé du nombre de CTC pour l’évaluation du pronostic chez les patients atteints de cancers métastasés de la prostate, du côlon-rectum et du sein à l’aide du dispositif CellSearch® (Veridex). Il s’agit du seul appareil approuvé par la FDA pour la détermination de la CTC.
La détection ou l’enrichissement des cellules tumorales circulantes reste un défi technologique : Dans 10 ml de sang total, il y a environ 50 milliards de globules rouges, 50 millions de globules blancs, mais seulement entre 0 et 100 CTC. En principe, il existe deux méthodes techniques pour détecter et enregistrer les CTC dans le sang. On peut utiliser pour cela une approche basée sur les propriétés biologiques des CTC, notamment sur l’expression de protéines présentes uniquement dans les CTC, mais pas dans les cellules sanguines. Ainsi, les CTC peuvent être capturés par des anticorps dirigés contre des protéines spécifiques des cellules épithéliales, telles que l’EpCAM ou les cytokératines. Une autre possibilité est de reconnaître les CTC en fonction de leurs différentes caractéristiques physiques, telles que la densité, la taille ou la déformabilité.
Diagnostic prédictif des tumeurs CTC pour l’avenir
Dans le passé, les essais cliniques se sont concentrés sur le nombre de CTC et la corrélation avec la survie. Grâce aux progrès technologiques, il est aujourd’hui possible d’extraire ces cellules et de les analyser à l’aide du NGS ou d’autres technologies, comme la PCR en temps réel [3]. Bien que cette approche soit actuellement principalement utilisée dans le cadre de la recherche, il existe des espoirs légitimes de pouvoir effectuer à l’avenir des diagnostics prédictifs de tumeurs sur ces cellules.
L’exemple le plus prometteur à ce jour d’une application prédictive de la CTC est la détermination du statut AR-V7 chez les patients atteints d’un cancer de la prostate résistant à la castration. AR-V7 est une variante d’épissage du gène du récepteur des androgènes, qui conduit à une protéine modifiée qui est constitutivement active. Antonarakis et al. ont montré que les patients dont les CTC exprimaient l’AR-V7 répondaient moins bien aux traitements par enzalutamide et abiratérone [4]. Le test utilisé dans cette étude est basé sur la capture des CTC à l’aide d’anticorps et la réalisation d’une PCR en temps réel avec des amorces AR-V7 spécifiques sur l’ARNm extrait. Ce test AR-V7 basé sur CTC sera commercialisé dans un avenir proche. Il pourrait être considéré comme une étape importante dans l’utilisation de la CTC pour détecter les mutations, les fusions de gènes et d’autres marqueurs prédictifs.
ADN acellulaire circulant (cfDNA)
Alors que les premières études sur le cfDNA ont examiné une éventuelle corrélation entre la quantité de cfDNA et le pronostic, il est aujourd’hui possible d’utiliser le NGS pour déterminer la séquence de bases du cfDNA (c.-à-d. le ctDNA) issu des cellules tumorales. Comme il n’est pas possible, pour des raisons techniques, de faire la différence entre l’ADNc général et l’ADNtc lors de l’extraction de l’ADNc, il se peut que la majeure partie de l’ADNc soit constituée d’ADNcn et non d’ADNtc. Pour pouvoir néanmoins déterminer les mutations dans l’ADNc, le NGS doit atteindre une grande profondeur. Pour les études sur l’ADNcf, le plasma sanguin est nécessaire.
Le NGS a récemment permis de détecter les mutations de la tumeur dans l’ADNc chez jusqu’à 75% des patients atteints de tumeurs avancées [5]. Pour les tumeurs non avancées, le taux de détection tombe à environ 50%. On peut toutefois s’attendre à ce que ces taux augmentent grâce à l’utilisation de méthodes plus sensibles.
Plusieurs études ont montré que la quantité d’ADNc est corrélée à la charge tumorale : si la tumeur répond au traitement, la quantité d’ADNc dans le sang du patient diminue. L’inverse se produit lors de la survenue d’une récidive : sur un petit collectif de patientes atteintes d’un cancer du sein, Dawson et al. montrer une augmentation des taux d’ADNc dans le sang des semaines avant que l’imagerie standard ne puisse détecter la récidive [6]. Pour ces examens, il suffit de déterminer ou de quantifier dans le sang quelques mutations spécifiques à la tumeur en question. Si l’on souhaite utiliser la biopsie liquide pour déterminer si la tumeur d’un patient présente une mutation prédictive, on utilise généralement pour le séquençage ce que l’on appelle des panels, dont la taille varie fortement (fig. 2).
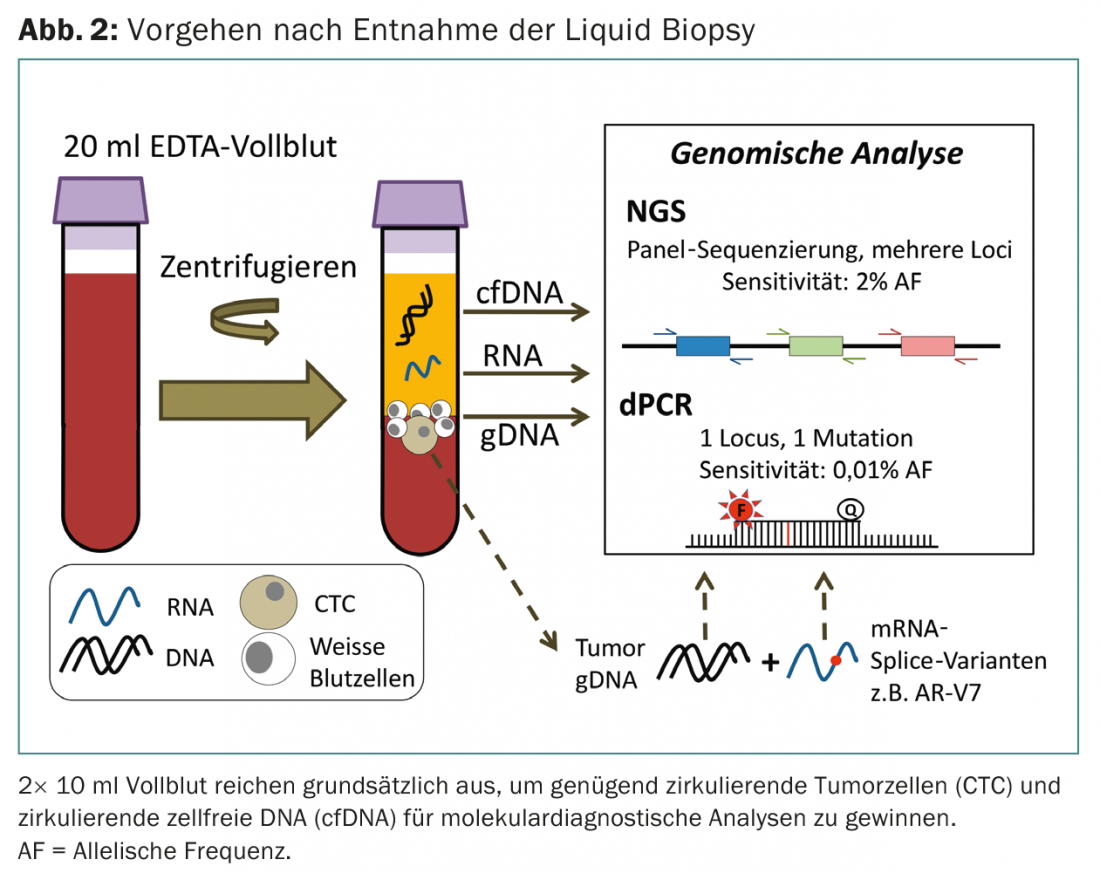
Les panels utilisés de manière standard dans le diagnostic des tumeurs couvrent des mutations entre 20 et 50 gènes. Ces gènes sont des gènes pertinents pour la thérapie, tels que EGFR, KRAS, NRAS ou BRAF. Comme nous l’avons dit, le séquençage de l’ADNc nécessite des profondeurs de séquençage élevées, les ressources et les coûts de séquençage augmentent avec la taille du panel.
En raison de la grande pertinence clinique des applications, les analyses basées sur le cfDNA sont désormais très proches de la pratique clinique quotidienne : le service de pathologie de l’Hôpital universitaire de Bâle propose depuis avril de cette année l’analyse du cfDNA pour le diagnostic. Il s’agit d’analyser l’ADNcf des patients à l’aide de panels NGS ou de PCR numérique afin de détecter les mutations ayant des propriétés prédictives. Ceci est particulièrement intéressant dans le cas où une biopsie tissulaire n’est pas possible ou raisonnablement possible chez un patient. Cette application est actuellement introduite principalement dans le diagnostic prédictif du cancer du poumon. Une extension à d’autres entités tumorales est toutefois prévisible.
Les analyses cfDNA permettent de déterminer de manière non invasive les mutations EGFR connues et pertinentes pour le traitement, en particulier la mutation EGFR T790M, qui est prédictive de la réponse à l’osimertinib chez les patients résistants aux inhibiteurs de la tyrosine kinase de l’EGFR (ITK-EGFR).
Un cas pratique
Une mutation du gène EGFR (p.E746-T751delinsF) a été détectée il y a trois ans par séquençage de cellules tumorales issues d’un lavage broncho-alvéolaire chez une patiente atteinte d’un adénocarcinome pulmonaire de stade IV positif pour le TTF1. La patiente a ensuite été traitée pendant huit mois par l’erlotinib, un EGFR-TKI, puis, en deuxième intention, par l’afatinib (fig. 3).
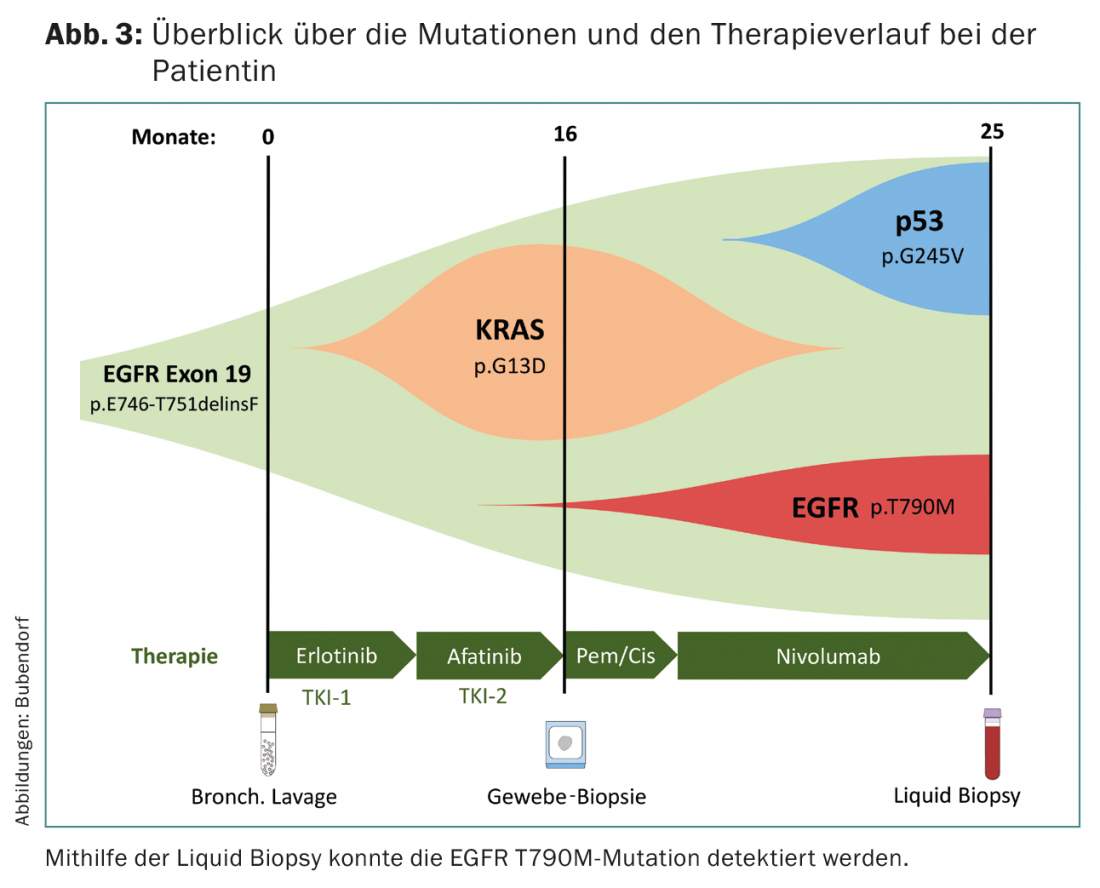
Le séquençage d’une biopsie tissulaire a révélé une mutation KRAS (p.G13D) qui suggère une résistance à l’EGFR-TKI. La patiente est passée à une chimiothérapie de courte durée, suivie d’un traitement par nivolumab, un inhibiteur de PD 1, en raison d’effets secondaires importants de l’EGFR-TKI. L’imagerie montrait à ce moment-là une progression de la tumeur. En raison de l’inaccessibilité de la tumeur, une biopsie liquide a été réalisée : En plus de la mutation EGFR initiale, une nouvelle mutation TP53 et une mutation EGFR T790M ont été détectées. Cette dernière est prédictive de la réponse à l’osimertinib. La patiente a été traitée par osimertinib et a présenté une réponse partielle à la tomodensitométrie deux mois après le début du traitement.
Littérature :
- Cheung KJ, et al. : A collective route to metastasis : Seeding by tumor cell clusters. Science 2016 Avril 8 ; 352(6282) : 167-169.
- De Bono JS, et al : Les cellules tumorales circulantes prédisent le bénéfice en termes de survie du traitement du cancer de la prostate métastatique résistant à la castration. Clin Cancer Res 2008 Oct 1 ; 14(19) : 6302-6309.
- Lohr JG, et al : Whole-exome sequencing of circulating tumor cells provides a window into metastatic prostate cancer. Nat Biotechnol 2014 May ; 32(5) : 479-484.
- Antonarakis ES, et al : AR-V7 et résistance à l’enzalutamide et à l’abiraterone dans le cancer de la prostate. N Engl J Med 2014 Sep 11 ; 371(11) : 1028-1038.
- Bettegowda C, et al : Détection de l’ADN tumoral circulant dans les tumeurs humaines précoces et tardives. Sci Transl Med 2014 Feb 19 ; 6(224) : 224ra24.
- Dawson SJ, et al : Analyse de l’ADN tumoral circulant pour surveiller le cancer du sein métastatique. N Engl J Med 2013 Mar 28 ; 368(13) : 1199-1209.
InFo ONKOLOGIE & HÉMATOLOGIE 2016 ; 4(4) : 10-13