Nella diagnostica tumorale, il termine biopsie liquide si riferisce all’analisi del DNA libero da cellule circolanti (cfDNA) e delle cellule tumorali circolanti. Oggi è possibile rilevare le mutazioni di un tumore in fase avanzata in modo minimamente invasivo, utilizzando le analisi del cfDNA. Il plasma, non il siero, deve essere utilizzato per il test del cfDNA. Le cellule tumorali circolanti potranno essere utilizzate per l’analisi dei marcatori predittivi nel prossimo futuro, come ad esempio per la determinazione della variante di splicing AR-V7 nel carcinoma prostatico resistente alla castrazione.
Sebbene il termine biopsie liquide sia relativamente nuovo nel contesto della diagnostica molecolare dei tumori, è diventato ampiamente accettato dai patologi e dagli oncologi. Nella diagnostica tumorale, questo termine è stato utilizzato inizialmente per l’analisi delle cellule tumorali circolanti (CTC) e solo successivamente per il DNA libero da cellule circolanti (cfDNA). In sintesi, si tratta di un termine generico per indicare il prelievo di materiale da un tumore solido mediante un semplice prelievo di sangue. Tuttavia, il termine “biopsia liquida” non è limitato esclusivamente all’uso nella diagnostica tumorale: Nella diagnostica prenatale, i campioni di sangue possono essere utilizzati per trarre conclusioni sulle anomalie cromosomiche del feto.
Interesse per la diagnosi minimamente invasiva
Le cellule tumorali circolanti (CTC) e il DNA libero da cellule circolanti (cfDNA) sono stati studiati per decenni e discussi in letteratura come possibili marcatori diagnostici e prognostici. La prima descrizione della CTC avvenne nel XIX secolo ad opera dell’australiano J. Ashworth (1869), quella del cfDNA poco meno di un secolo dopo ad opera dei due francesi P. Mandel e P. Metais (1948).
Le CTC possono provenire dal tumore primario e dalle metastasi. Non è ancora chiaro se il rilascio di CTC sia un processo attivo e quindi parte di un programma biologico di un tumore, o se il rilascio avvenga in modo casuale. Tuttavia, è noto che alcune di queste cellule hanno la capacità di sopravvivere nel flusso sanguigno e di continuare a moltiplicarsi in altre parti del corpo umano. per formare una metastasi. È controverso se siano sufficienti singole cellule per questo o se le associazioni (cluster) di cellule tumorali non siano responsabili del processo di metastasi ematogena a distanza [1].
Il DNA libero da cellule circolanti (cfDNA), invece, proviene da cellule morenti (apoptotiche o necrotiche). Il cfDNA è costituito dal DNA delle cellule tumorali (DNA tumorale libero da cellule circolanti, abbreviato ctDNA) e delle cellule sane (DNA normale libero da cellule circolanti, abbreviato cnDNA). È controverso se il cfDNA nel sangue svolga una funzione. Rispetto alla CTC (1-2,5 ore), il cfDNA ha un’emivita relativamente breve di 15-30 minuti.
Sia le CTC che il cfDNA/ctDNA stanno acquisendo grande importanza per la diagnostica dei tumori, grazie alle nuove possibilità di indagini genomiche. Oggi, il sequenziamento di nuova generazione (NGS) consente di sequenziare interi genomi in tempi molto brevi. Dopo l’uso di questa tecnologia in un gran numero di studi clinici, il numero di marcatori genomici potenzialmente predittivi è aumentato notevolmente e di conseguenza l’interesse per il rilevamento minimamente invasivo di questi marcatori predittivi nel sangue.
Determinare le mutazioni in modo significativo
Rispetto al sequenziamento Sanger convenzionale, l’NGS consente di sequenziare simultaneamente regioni più ampie e soprattutto con una profondità elevata (ridondanza). Questa profondità è necessaria se si vogliono rilevare mutazioni subclonali o se il DNA tumorale rappresenta solo una frazione del DNA totale da analizzare. Quest’ultimo è il caso dell’analisi del ctDNA: a seconda dell’entità del tumore e dello stadio della malattia tumorale, il contenuto di ctDNA nel sangue può variare notevolmente o ammontare fino a pochi per mille.
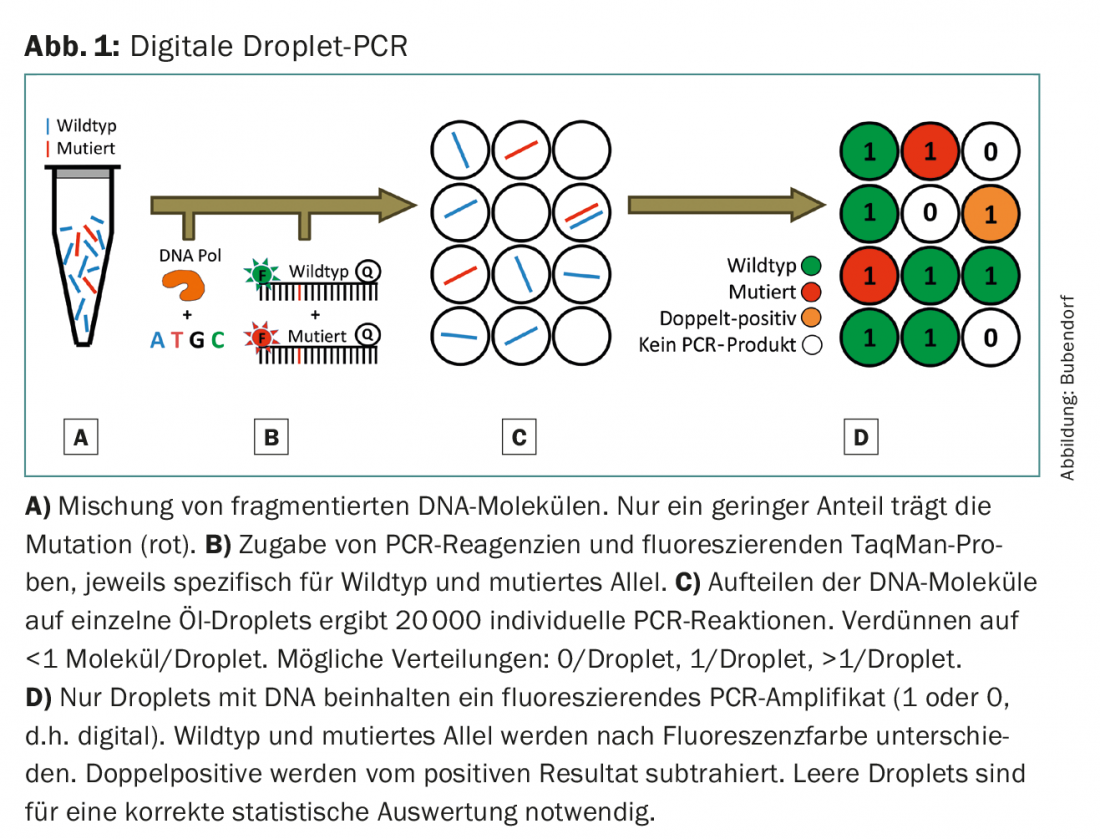
Con la maggior parte dei sequenziatori NGS, i protocolli standard possono rilevare mutazioni che rappresentano solo il 2-5% del DNA totale. Tuttavia, il ctDNA è spesso inferiore a questa soglia, soprattutto nei tumori non ancora avanzati o nei pazienti in terapia. A differenza della NGS, la PCR digitale permette di determinare una mutazione in questo intervallo altamente sensibile: con l’aiuto di sonde specifiche marcate con fluorescenza, alcune mutazioni possono essere determinate in modo significativo nell’intervallo dello 0,01%. La procedura e la tecnica sono descritte con l’esempio della PCR digitale a gocce (Fig. 1). Lo svantaggio di questo metodo è che deve essere effettuata una reazione separata per ogni mutazione da determinare. Pertanto, non è consigliabile utilizzare questa metodologia per un’indagine generale del genoma, ma solo per l’indagine di singole mutazioni.
Cellule tumorali circolanti (CTC)
Nell’ultimo decennio, ci sono stati diversi studi che hanno dimostrato che il rilevamento di una maggiore quantità di CTC nel sangue dei pazienti con cancro avanzato è associato a una scarsa sopravvivenza [2]. In questo contesto, l’autorità regolatoria americana FDA ha approvato la raccolta del numero di CTC per la valutazione della prognosi nei pazienti con tumore metastatico della prostata, del colon-retto e del seno, utilizzando il dispositivo CellSearch® (Veridex). Questo è l’unico dispositivo approvato dalla FDA per la determinazione delle CTC.
Il rilevamento o l’arricchimento delle cellule tumorali circolanti rimane una sfida tecnologica: In 10 ml di sangue intero ci sono circa 50 miliardi di globuli rossi, 50 milioni di globuli bianchi, ma solo tra 0 e 100 CTC. Fondamentalmente, esistono due metodi tecnici per rilevare e registrare le CTC nel sangue. A tal fine, si può utilizzare un approccio che si basa sulle proprietà biologiche delle CTC, in particolare sull’espressione di proteine che si trovano solo nelle CTC ma non nelle cellule del sangue. Quindi, le CTC possono essere catturate con anticorpi contro proteine specifiche delle cellule epiteliali, come EpCAM o citocheratine. Un’altra possibilità è quella di riconoscere le CTC in base alle loro diverse proprietà fisiche, come la densità, le dimensioni o la deformabilità.
Diagnosi predittiva dei tumori CTC per il futuro
In passato, gli studi clinici si sono concentrati sul numero di CTC e sulla correlazione con la sopravvivenza. Grazie ai progressi tecnologici, oggi è possibile pescare queste cellule e analizzarle utilizzando l’NGS o altre tecnologie, come la PCR in tempo reale [3]. Sebbene questo approccio sia attualmente utilizzato principalmente nella ricerca, ci sono speranze fondate che in futuro si possa eseguire una diagnostica predittiva del tumore su queste cellule.
L’esempio più promettente di uso predittivo delle CTC, ad oggi, è la determinazione dello stato di AR-V7 nei pazienti con cancro alla prostata resistente alla castrazione. AR-V7 è una variante di splicing del gene del recettore degli androgeni che porta a una proteina alterata che è costitutivamente attiva. Antonarakis et al. hanno potuto dimostrare che i pazienti le cui CTC esprimevano l’RNA AR-V7 rispondevano peggio alle terapie con enzalutamide e abiraterone [4]. Il test utilizzato in questo studio si basa sulla cattura delle CTC mediante anticorpi e sull’esecuzione della PCR in tempo reale con primer specifici per AR-V7 sull’mRNA estratto. Questo test AR-V7 basato sulle CTC sarà commercializzato nel prossimo futuro. Potrebbe essere considerata una pietra miliare per l’uso delle CTC per rilevare mutazioni, fusioni geniche e altri marcatori predittivi.
DNA libero da cellule circolanti (cfDNA)
Mentre i primi studi sul cfDNA hanno studiato una possibile correlazione tra la quantità di cfDNA e la prognosi, oggi la NGS può essere utilizzata per determinare la sequenza di basi del cfDNA (cioè il ctDNA) proveniente dalle cellule tumorali. Poiché la differenziazione tra cfDNA generale e ctDNA non è possibile per motivi tecnici quando si estrae il cfDNA, la maggior parte del cfDNA può essere costituita da cnDNA e non da ctDNA. Tuttavia, per determinare le mutazioni nel ctDNA, è necessario raggiungere un’elevata profondità con la NGS. Per il test del cfDNA è necessario il plasma sanguigno.
Utilizzando la NGS, recentemente è stato possibile rilevare le mutazioni del tumore nel ctDNA di fino al 75% dei pazienti con tumori avanzati [5]. Nei tumori non avanzati, il tasso di rilevamento scende a circa il 50%. Tuttavia, si può ipotizzare che questi tassi aumenteranno grazie all’uso di metodi più sensibili.
Diversi studi hanno dimostrato che la quantità di ctDNA è correlata al carico tumorale: se il tumore risponde alla terapia, la quantità di ctDNA nel sangue del paziente diminuisce. Il contrario è vero per il verificarsi di una recidiva: in un piccolo gruppo di pazienti con cancro al seno, Dawson et al. mostrano un aumento dei livelli di ctDNA nel sangue settimane prima che la diagnostica per immagini standard possa rilevare la recidiva [6]. Per questi esami, è sufficiente determinare o quantificare alcune mutazioni nel sangue specifiche del tumore corrispondente. Se si vuole utilizzare la biopsia liquida per indagare se il tumore di un paziente presenta una mutazione predittiva, di solito si utilizzano i cosiddetti pannelli per il sequenziamento, che variano molto in termini di dimensioni (Fig. 2).
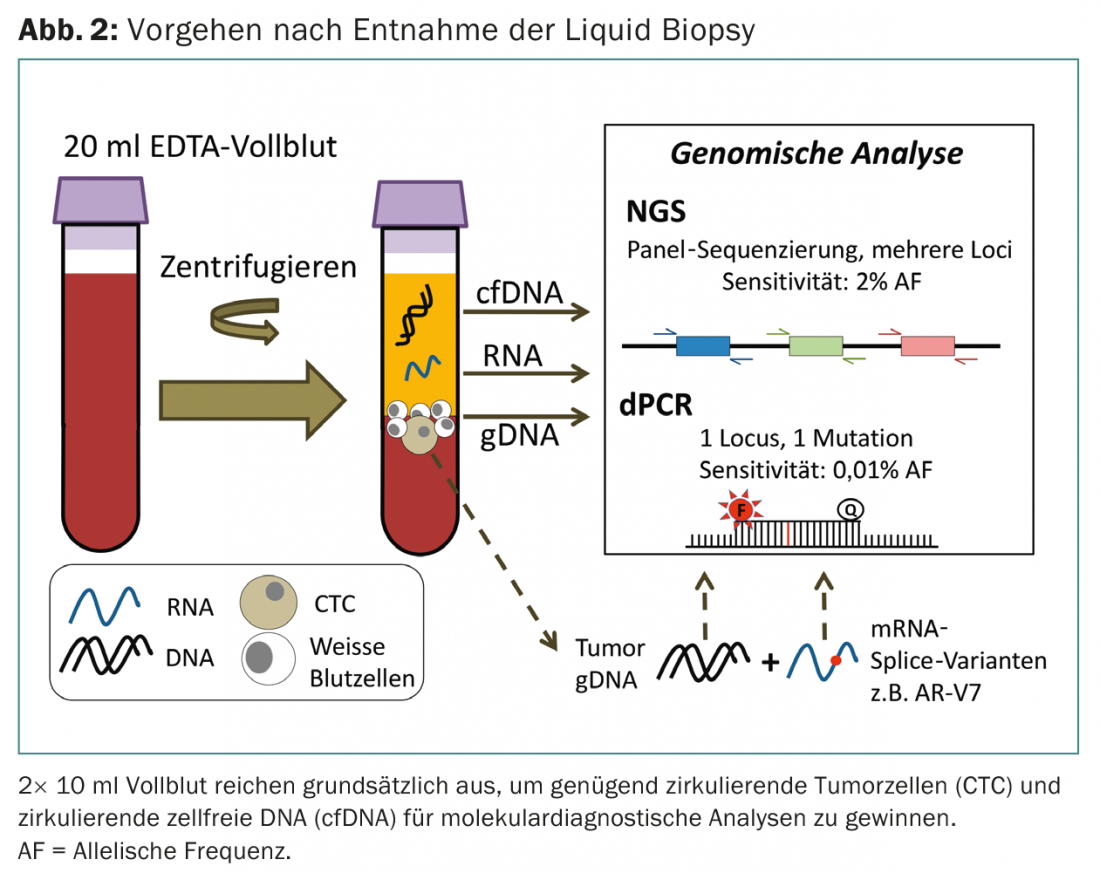
I pannelli standard utilizzati nella diagnostica dei tumori coprono le mutazioni di 20-50 geni. Questi geni sono geni rilevanti per la terapia, come EGFR, KRAS, NRAS o BRAF. Poiché, come detto, per il sequenziamento del cfDNA sono necessarie profondità di sequenziamento elevate, le risorse e i costi di sequenziamento aumentano con le dimensioni del pannello.
Grazie all’elevata rilevanza clinica delle applicazioni, gli esami basati sul cfDNA sono già molto vicini alla pratica clinica quotidiana: il reparto di patologia dell’Ospedale Universitario di Basilea offre analisi del cfDNA per la diagnostica dall’aprile di quest’anno. In questo processo, il cfDNA dei pazienti viene analizzato per le mutazioni con proprietà predittive utilizzando pannelli NGS o la PCR digitale. Questo è di particolare interesse se una biopsia tissutale non è possibile o non è ragionevole per un paziente. Questa applicazione viene attualmente introdotta soprattutto nella diagnosi predittiva dei carcinomi polmonari. Tuttavia, è prevedibile un’espansione ad altre entità tumorali.
Mediante esami del cfDNA, è possibile determinare in modo non invasivo le mutazioni EGFR note e rilevanti per la terapia, in particolare anche la mutazione EGFR T790M, che è predittiva della risposta a osimertinib nei pazienti con resistenza agli inibitori della tirosin-chinasi EGFR (EGFR-TKI).
Un caso pratico
Tre anni fa, è stata rilevata una mutazione nel gene EGFR (p.E746-T751delinsF) in un paziente con un adenocarcinoma del polmone TTF1-positivo, in stadio IV, mediante il sequenziamento delle cellule tumorali di un lavaggio broncoalveolare. Il paziente è stato poi trattato con l’EGFR TKI erlotinib per otto mesi e successivamente, come terapia di seconda linea, con afatinib (Fig. 3).
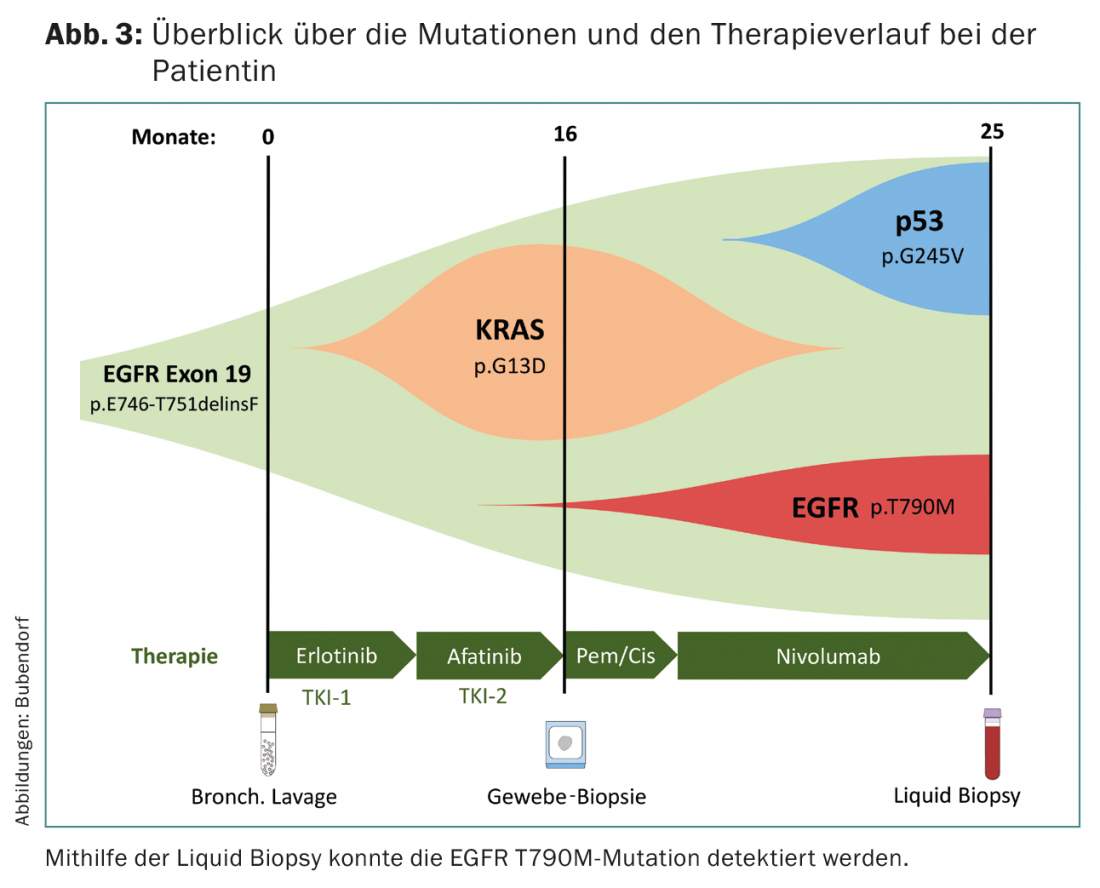
Il sequenziamento di una biopsia tissutale ha rivelato una mutazione KRAS (p.G13D) suggestiva di resistenza agli EGFR-TKI. Il paziente è passato alla chemioterapia a breve termine a causa dei gravi effetti collaterali dei TKI EGFR, seguita dal trattamento con l’inibitore PD 1 nivolumab. La diagnostica per immagini ha mostrato una progressione del tumore in questo periodo. A causa dell’inaccessibilità del tumore, è stata eseguita una biopsia liquida: Oltre alla mutazione EGFR originale, sono state rilevate una mutazione TP53 e una mutazione EGFR T790M. Quest’ultimo è predittivo della risposta a osimertinib. Il paziente è stato trattato con osimertinib e ha mostrato una risposta parziale nella tomografia computerizzata due mesi dopo l’inizio della terapia.
Letteratura:
- Cheung KJ, et al.: Una via collettiva alla metastasi: la semina da parte di gruppi di cellule tumorali. Science 2016 Apr 8; 352(6282): 167-169.
- De Bono JS, et al: Le cellule tumorali circolanti predicono il beneficio di sopravvivenza dal trattamento nel cancro alla prostata metastatico resistente alla castrazione. Clin Cancer Res 2008 Oct 1; 14(19): 6302-6309.
- Lohr JG, et al: Il sequenziamento dell’intero esoma delle cellule tumorali circolanti fornisce una finestra sul cancro alla prostata metastatico. Nat Biotechnol 2014 maggio; 32(5): 479-484.
- Antonarakis ES, et al: AR-V7 e resistenza a enzalutamide e abiraterone nel cancro alla prostata. N Engl J Med 2014 Sep 11; 371(11): 1028-1038.
- Bettegowda C, et al: Rilevamento del DNA tumorale circolante nei tumori umani in fase iniziale e avanzata. Sci Transl Med 2014 Feb 19; 6(224): 224ra24.
- Dawson SJ, et al: Analisi del DNA tumorale circolante per monitorare il cancro al seno metastatico. N Engl J Med 2013 Mar 28; 368(13): 1199-1209.
InFo ONCOLOGIA ED EMATOLOGIA 2016; 4(4): 10-13