No diagnóstico de tumores, o termo biopsias líquidas refere-se à análise de ADN livre de células circulantes (cfDNA) e células tumorais circulantes. Actualmente, é possível detectar as mutações de um tumor em tumores avançados de uma forma minimamente invasiva, utilizando análises cfDNA. O plasma, não o soro, deve ser utilizado para testes de cfDNA. As células tumorais circulantes poderão ser utilizadas para análise de marcadores preditivos num futuro próximo, tal como para a determinação da variante de emenda de AR-V7 no carcinoma da próstata resistente à castração.
Embora o termo biópsias líquidas seja relativamente novo no contexto do diagnóstico de tumores moleculares, tornou-se amplamente aceite entre patologistas e oncologistas. No diagnóstico de tumores, este termo foi primeiro utilizado para a análise de células tumorais circulantes (CTC) e só mais tarde para ADN livre de células circulantes (cfDNA). Em resumo, este é um termo genérico para a remoção de material de um tumor sólido através da simples recolha de uma amostra de sangue. No entanto, o termo “biopsia líquida” não se limita exclusivamente ao uso em diagnósticos de tumores: No diagnóstico pré-natal, as amostras de sangue podem ser utilizadas para tirar conclusões sobre anomalias cromossómicas no feto.
Interesse na detecção minimamente invasiva
As células tumorais circulantes (CTC) e o ADN sem células circulantes (cfDNA) têm sido pesquisados durante décadas e discutidos como possíveis marcadores de diagnóstico e prognóstico na literatura. A primeira descrição do CTC teve lugar no século XIX pelo australiano J. Ashworth (1869), a do cfDNA um pouco menos de um século depois pelos dois franceses P. Mandel e P. Metais (1948).
O CTC pode ter origem no tumor primário, bem como nas metástases. Ainda não é claro se a libertação de CTC é um processo activo e, portanto, parte de um programa biológico de um tumor, ou se a libertação ocorre de forma aleatória. No entanto, sabe-se que algumas destas células têm a capacidade de sobreviver na corrente sanguínea e continuam a multiplicar-se noutros locais do corpo humano. para formar uma metástase. Discute-se de forma controversa se células únicas são suficientes para isso ou se as associações (aglomerados) de células tumorais não são responsáveis pelo processo de metástase hematogénica à distância [1].
O ADN circulante sem células (cfDNA), por outro lado, provém de células moribundas (apoptóticas ou necróticas). O cfDNA é constituído por ADN de células tumorais (ADN tumoral sem células circulantes, ctDNA abreviado) e células saudáveis (ADN normal sem células circulantes, cnDNA abreviado). É controverso se o cfDNA no sangue desempenha uma função. Em comparação com o CTC (1-2,5 horas), cfDNA tem uma meia-vida relativamente curta de 15-30 minutos.
Tanto o CTC como o cfDNA/ctDNA estão a ganhar forte importância para o diagnóstico de tumores graças às novas possibilidades de investigações genómicas. Actualmente, a Next Generation Sequencing (NGS) permite a sequenciação de genomas inteiros num espaço de tempo muito curto. Desde a utilização desta tecnologia num grande número de ensaios clínicos, o número de marcadores genómicos potencialmente preditivos tem aumentado acentuadamente e, consequentemente, o interesse na detecção minimamente invasiva destes marcadores preditivos no sangue.
Determinar as mutações de forma significativa
Em comparação com a sequenciação Sanger convencional, o NGS permite que regiões maiores sejam sequenciadas simultaneamente e especialmente com uma grande profundidade (redundância). Esta profundidade é necessária se se quiser detectar mutações subclonais ou se o ADN do tumor representar apenas uma fracção do ADN total a ser analisado. Este último é o caso da análise do ctDNA: Dependendo da entidade tumoral e da fase da doença tumoral, o conteúdo de ctDNA no sangue pode variar muito ou atingir até alguns por mililitro.
Com a maioria dos sequenciadores NGS, os protocolos padrão podem detectar mutações que representam apenas 2-5% do ADN total. No entanto, o ctDNA está frequentemente abaixo deste limiar, especialmente em tumores ainda não avançados ou em pacientes submetidos a terapia. Em contraste com o NGS, a PCR digital permite a determinação de uma mutação nesta gama altamente sensível: com a ajuda de sondas específicas com rótulo fluorescente, certas mutações podem ser determinadas significativamente na gama de 0,01%. O procedimento e técnica são descritos utilizando o exemplo da PCR digital de gotículas (Fig. 1) . A desvantagem deste método é que deve ser realizada uma reacção separada para cada mutação a ser determinada. Não se recomenda, portanto, a utilização desta metodologia para uma investigação geral do genoma, mas apenas para a investigação de mutações únicas.
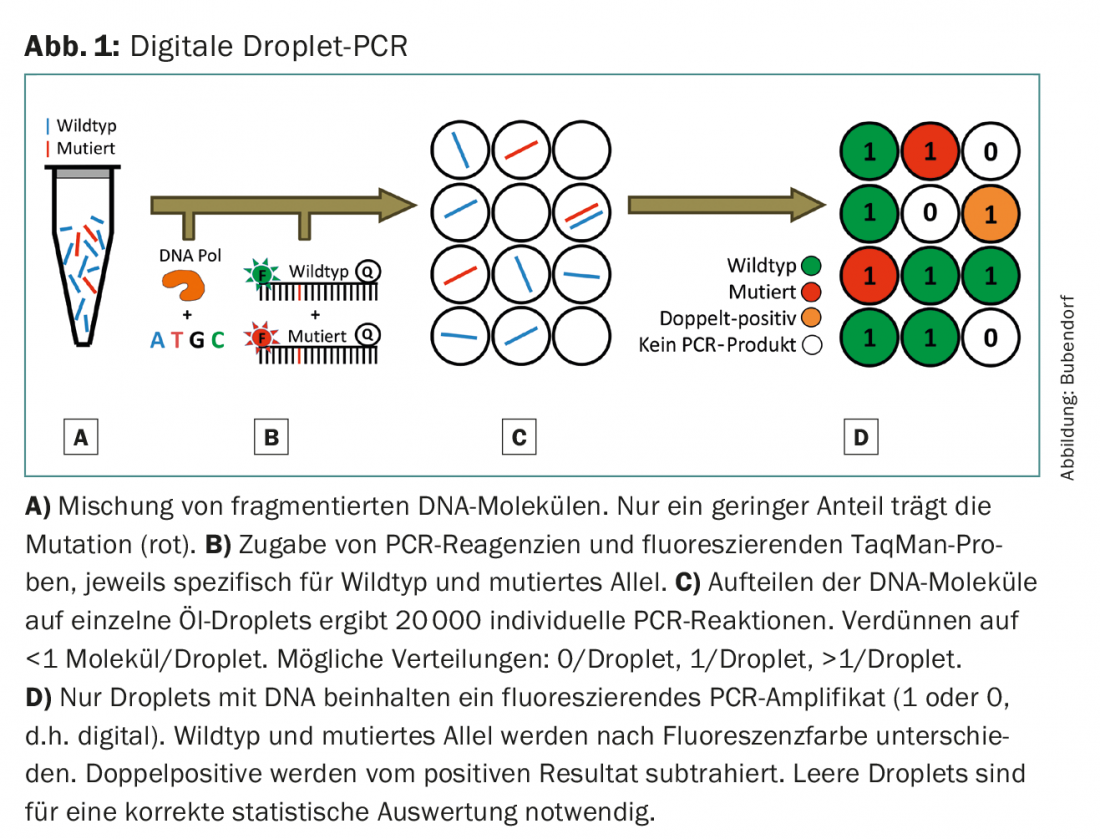
Células tumorais circulantes (CTC)
Na última década, houve uma série de estudos que mostram que a detecção de uma maior quantidade de CTC no sangue de doentes com cancro avançado está associada à fraca sobrevivência [2]. Neste contexto, a autoridade reguladora americana FDA aprovou a recolha do número de CTC para a avaliação do prognóstico em pacientes com cancro da próstata metastásico, colorrectal e da mama utilizando o dispositivo CellSearch® (Veridex). Este é o único dispositivo aprovado pela FDA para a determinação do CTC.
A detecção ou enriquecimento de células tumorais circulantes continua a ser um desafio tecnológico: Em 10 ml de sangue total existem cerca de 50 mil milhões de glóbulos vermelhos, 50 milhões de glóbulos brancos, mas apenas entre 0 e 100 CTC. Basicamente, existem dois métodos técnicos para detectar e registar o CTC no sangue. Pode-se utilizar para isso uma abordagem baseada nas propriedades biológicas do CTC, em particular na expressão de proteínas que só se encontram no CTC mas não nas células sanguíneas. Assim, o CTC pode ser capturado com anticorpos contra proteínas específicas de células epiteliais, tais como EpCAM ou citoceratinas. Outra possibilidade é reconhecer o CTC com base nas suas diferentes propriedades físicas, tais como densidade, tamanho ou deformabilidade.
Diagnóstico preditivo de tumores CTC para o futuro
No passado, os ensaios clínicos centravam-se no número de CTC e na correlação com a sobrevivência. Devido ao progresso tecnológico, é agora possível pescar estas células e analisá-las usando NGS ou outras tecnologias, tais como a PCR em tempo real [3]. Embora esta abordagem seja actualmente utilizada principalmente na investigação, há esperanças justificadas de que o diagnóstico preditivo de tumores possa ser realizado nestas células no futuro.
O exemplo mais promissor de utilização preditiva de CTC até à data é a determinação do estado AR-V7 em doentes com cancro da próstata resistente à castração. AR-V7 é uma variante de emenda do gene receptor androgénio que leva a uma proteína alterada que é constitutivamente activa. Antonarakis et al. foram capazes de mostrar que os doentes cujo CTC expressou o AR-V7 RNA reagiram pior às terapias com enzalutamida e abiraterona [4]. O ensaio utilizado neste estudo baseia-se na captura do CTC usando anticorpos e realizando PCR em tempo real com iniciadores AR-V7 específicos sobre o mRNA extraído. Este teste AR-V7 baseado no CTC será comercializado num futuro próximo. Pode ser considerado um marco para o uso de CTC para detectar mutações, fusões de genes e outros marcadores preditivos.
ADN sem células circulantes (cfDNA)
Enquanto os primeiros estudos de cfDNA investigaram uma possível correlação da quantidade de cfDNA com o prognóstico, actualmente o NGS pode ser utilizado para determinar a sequência base do cfDNA (ou seja, ctDNA) proveniente das células tumorais. Uma vez que a diferenciação entre cfDNA geral e ctDNA não é possível por razões técnicas ao extrair cfDNA, a maioria do cfDNA pode consistir em cnDNA e não em ctDNA. Para que ainda seja possível determinar as mutações no ctDNA, deve ser conseguida uma grande profundidade com NGS. O plasma sanguíneo é necessário para o teste de cfDNA.
Usando NGS, foi recentemente possível detectar as mutações do tumor no ctDNA de até 75% dos pacientes com tumores avançados [5]. Em tumores não avançados, a taxa de detecção cai para cerca de 50%. No entanto, pode assumir-se que estas taxas irão aumentar através da utilização de métodos mais sensíveis.
Vários estudos têm demonstrado que a quantidade de ctDNA está correlacionada com a carga tumoral: se o tumor responder à terapia, a quantidade de ctDNA no sangue do paciente diminui. O oposto é verdadeiro para a ocorrência de uma recorrência: num pequeno grupo de doentes com cancro da mama, Dawson et al. mostram um aumento dos níveis de ctDNA sanguíneo semanas antes que a imagem padrão pudesse detectar a recidiva [6]. Para estes exames, é suficiente determinar ou quantificar algumas mutações no sangue específicas do tumor correspondente. Se se quiser usar biopsia líquida para investigar se o tumor de um paciente tem uma mutação preditiva, os chamados painéis são normalmente usados para sequenciação, que variam muito em tamanho (Fig. 2).
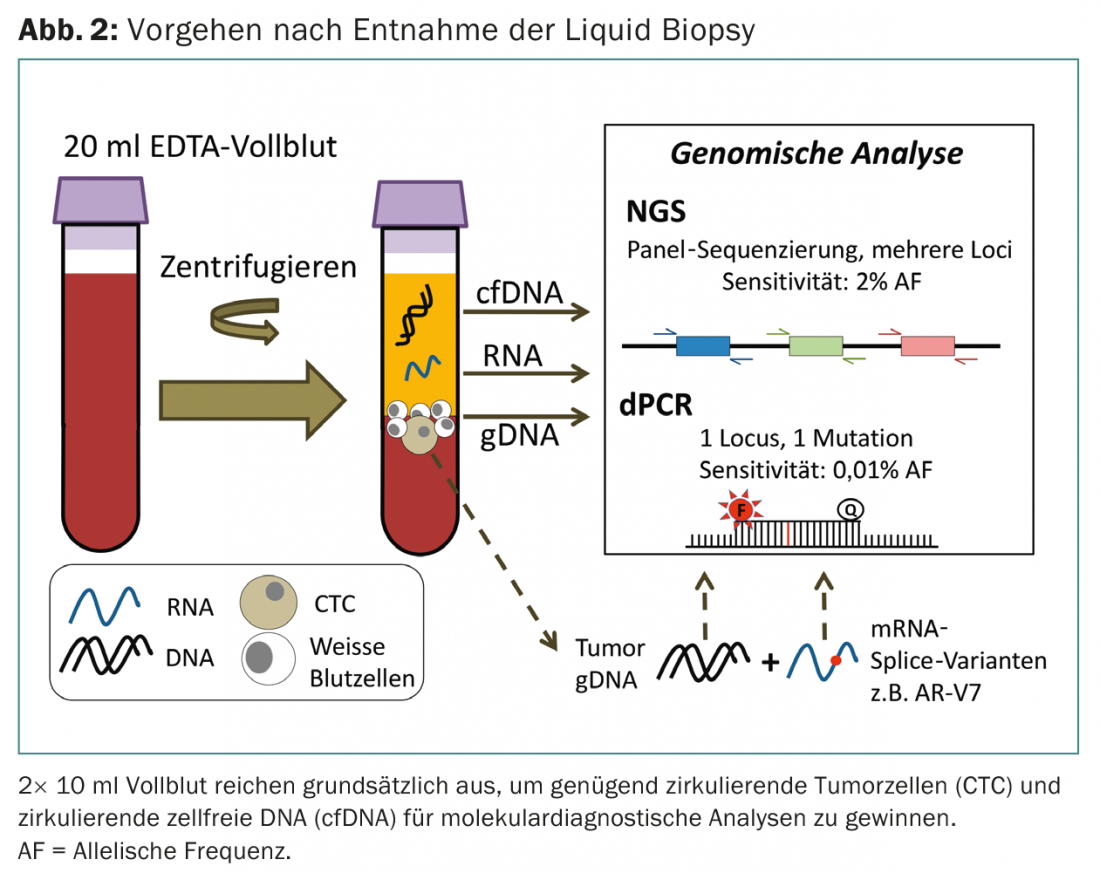
Os painéis padrão utilizados no diagnóstico de tumores cobrem mutações entre 20 e 50 genes. Estes genes são genes relevantes para a terapia, tais como EGFR, KRAS, NRAS ou BRAF. Uma vez que, tal como mencionado, são necessárias grandes profundidades de sequenciação para cfDNA, os recursos e custos de sequenciação aumentam com o tamanho do painel.
Devido à elevada relevância clínica das aplicações, os exames baseados em cfDNA já estão agora muito próximos da prática clínica diária: o departamento de patologia do Hospital Universitário de Basileia oferece análises cfDNA para diagnósticos desde Abril deste ano. Neste processo, o cfDNA dos pacientes é analisado para detectar mutações com propriedades preditivas utilizando painéis NGS ou PCR digital. Isto é de particular interesse se uma biópsia de tecido não for possível ou não for razoável para um paciente. Esta aplicação está actualmente a ser introduzida principalmente no diagnóstico preditivo de carcinomas pulmonares. No entanto, é previsível uma expansão para outras entidades tumorais.
Através de exames cfDNA, as mutações EGFR conhecidas e relevantes para a terapia podem ser determinadas de forma não invasiva, em particular também a mutação EGFR T790M, que é preditiva para a resposta ao osimertinib em pacientes com resistência aos inibidores de tirosina quinase EGFR (EGFR-TKIs).
Um caso da prática
Há três anos, foi detectada uma mutação no gene EGFR (p.E746-T751delinsF) num doente com um adenocarcinoma TTF1 positivo, estádio IV do pulmão, sequenciando células tumorais de um lavado broncoalveolar. O doente foi então tratado com o EGFR TKI erlotinib durante oito meses e subsequentemente, como terapia de segunda linha, com afatinibe (Fig. 3).
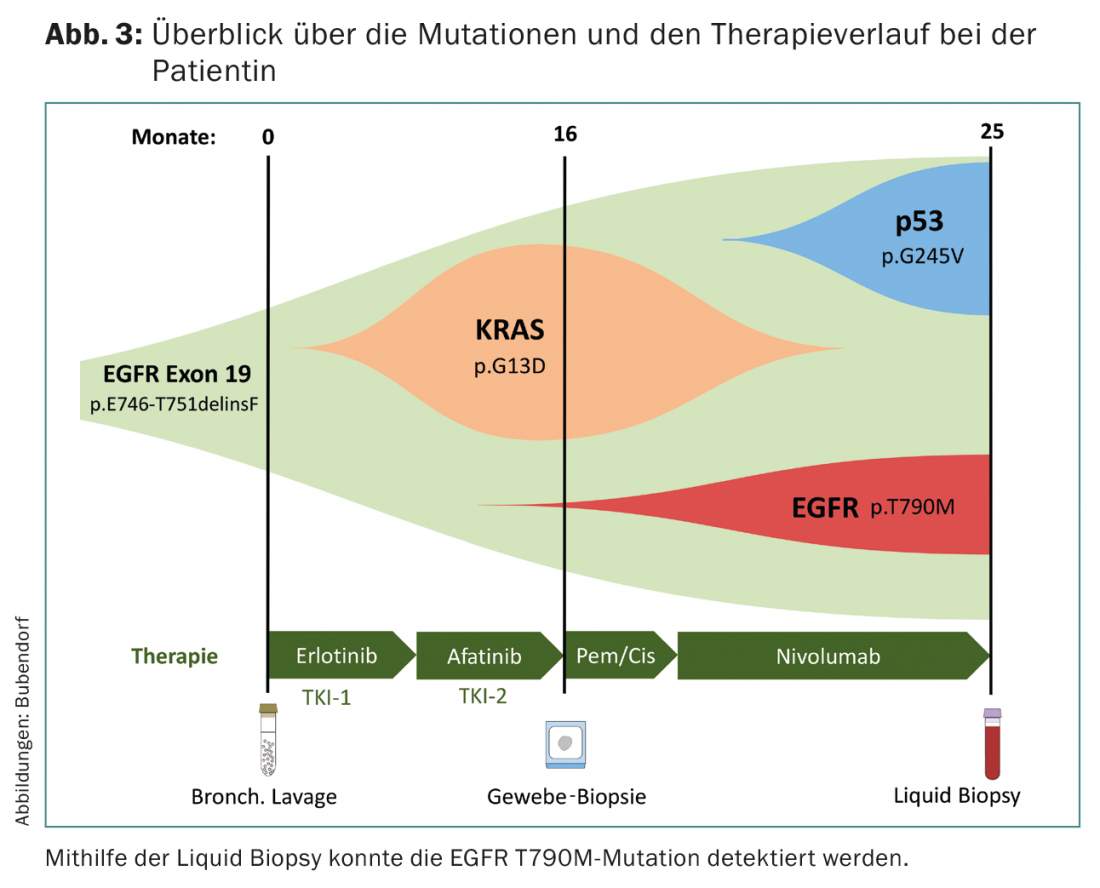
A sequência de uma biópsia de tecido revelou uma mutação KRAS (p.G13D) sugestiva de resistência a EGFR-TKIs. O paciente foi submetido a quimioterapia de curto prazo devido aos graves efeitos secundários das TKIs EGFR, seguido de tratamento com o inibidor PD 1 nivolumab. As imagens mostraram a progressão de tumores neste momento. Devido à inacessibilidade do tumor, foi realizada uma biópsia líquida: Além da mutação EGFR original, foram recentemente detectadas uma mutação TP53 e uma mutação EGFR T790M. Este último é preditivo de resposta ao osimertinib. O paciente foi tratado com osimertinibe e mostrou uma resposta parcial em tomografia computorizada dois meses após ter iniciado a terapia.
Literatura:
- Cheung KJ, et al.: Uma via colectiva para a metástase: Semeadura por grupos de células tumorais. Ciência 2016 Abril 8; 352(6282): 167-169.
- De Bono JS, et al: As células tumorais circulantes prevêem o benefício da sobrevivência do tratamento no cancro da próstata resistente à castração metastática. Clin Cancer Res 2008 Oct 1; 14(19): 6302-6309.
- Lohr JG, et al: A sequência completa de células tumorais circulantes proporciona uma janela para o cancro da próstata metastásico. Nat Biotechnol 2014 Maio; 32(5): 479-484.
- Antonarakis ES, et al: AR-V7 e resistência à enzalutamida e à abiraterona no cancro da próstata. N Engl J Med 2014 Set 11; 371(11): 1028-1038.
- Bettegowda C, et al: Detecção de ADN de tumores circulantes em fases iniciais e tardias de malignidade humana. Sci Transl Med 2014 Fev 19; 6(224): 224ra24.
- Dawson SJ, et al: Análise de ADN tumoral circulante para monitorizar o cancro da mama metastásico. N Engl J Med 2013 Mar 28; 368(13): 1199-1209.
InFo ONCOLOGy & HEMATOLOGy 2016; 4(4): 10-13