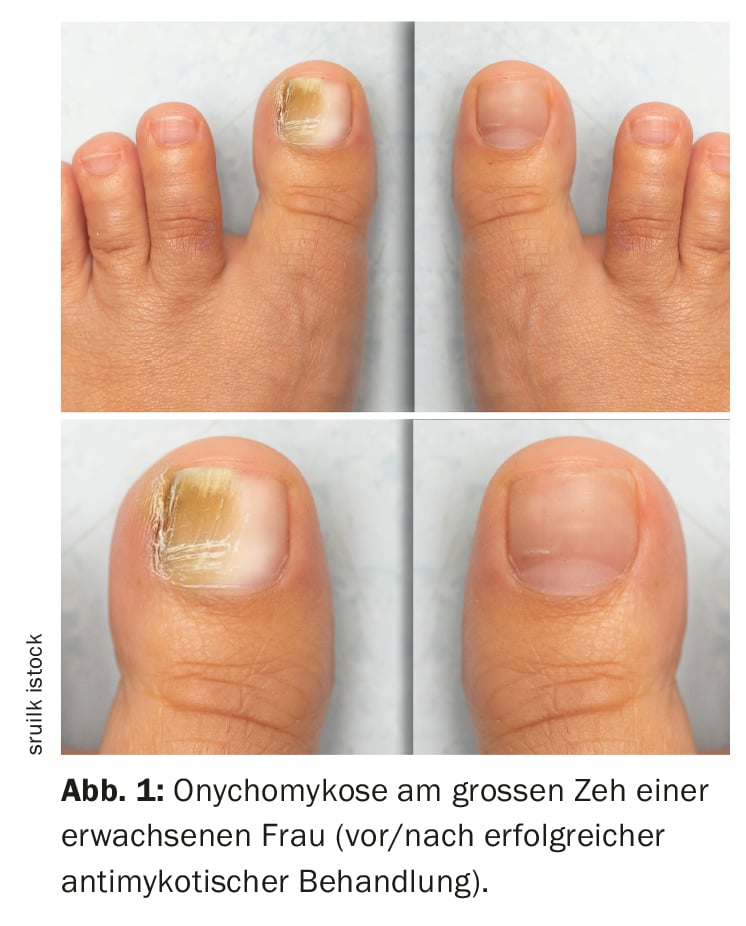
Un team di ricerca internazionale che ha coinvolto gli Ospedali Universitari di Losanna e Zurigo ha confrontato diversi metodi diagnostici in uno studio retrospettivo su larga scala. In linea con i risultati empirici precedenti, la conclusione dello studio è che i metodi molecolari sono superiori alla diagnostica fungina convenzionale quando si sospetta un’onicomicosi.
Con una prevalenza stimata del 4-9% nella popolazione generale, le onicomicosi sono la causa più comune di aspetto anomalo dell’apparato ungueale [1,2]. L’identificazione rapida e affidabile dell’agente patogeno è fondamentale per avviare la terapia appropriata il più rapidamente possibile. Rispetto ai metodi diagnostici convenzionali, i metodi molecolari sono più rapidi e hanno una maggiore precisione nell’identificazione delle specie. Gli strumenti diagnostici molecolari attualmente disponibili si differenziano principalmente per i metodi di estrazione del DNA, i primer PCR utilizzati e i prodotti dell’analisi PCR [3]. Rispetto ai metodi diagnostici convenzionali, i metodi molecolari sono più rapidi (risultati entro un giorno) e hanno una maggiore accuratezza, il che è molto interessante per l’applicazione nella pratica clinica [4].
Microscopia a fluorescenza e coltura fungina vs. sequenziamento PCR
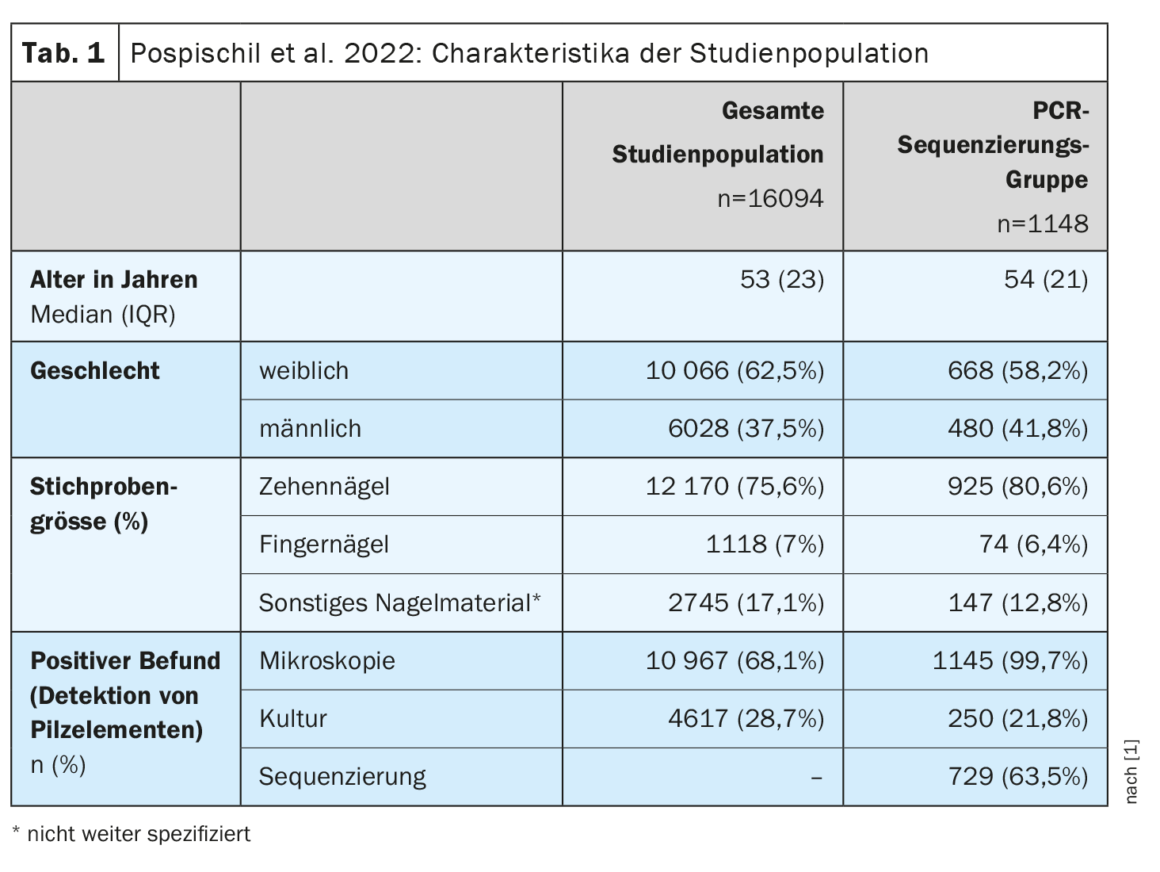
Un totale di 16.094 campioni di unghie sono stati esaminati con la microscopia a fluorescenza diretta [1]. Inoltre, due colture fungine sono state ottenute da ciascun campione utilizzando l’agar Sabouraud dextrose con cloramfenicolo (50 μg/mL) e cloramfenicolo più cicloeximide (400 μg/mL). Con la microscopia a fluorescenza diretta, sono stati rilevati elementi fungini nel 68,1% dei campioni di unghie; con la coltura fungina, il tasso di rilevamento è stato del 28,7%. I due metodi hanno mostrato una concordanza complessiva del 58,4%. La maggior parte dei risultati discordanti derivava da campioni con risultati positivi alla microscopia diretta ma negativi alla coltura fungina. In un totale del 59,5% dei campioni in cui sono stati rilevati elementi fungini mediante microscopia a fluorescenza, la coltura fungina è risultata negativa. Tra i pazienti con un risultato positivo nella coltura fungina, i dermatofiti rappresentavano la percentuale maggiore (47,9%), con Trichophyton rubrum come patogeno più comune, seguito da “muffe non dermatofite” (NDM) (29,1%), lieviti (21,8%) e infezioni miste (1,2%). In 1148 casi, la diagnostica fungina convenzionale mediante microscopia a fluorescenza e coltura fungina si è rivelata insufficiente, motivo per cui è stato utilizzato un ulteriore chiarimento diagnostico molecolare (Tab. 1) [1].
La coltura fungina ha rilevato i patogeni fungini solo nel 21,8% dei casi e quindi ha mostrato una bassa sensibilità, mentre il sequenziamento PCR si è dimostrato nettamente superiore con un tasso di rilevamento del 63,5%. La distribuzione di frequenza dei diversi elementi fungini identificati mediante sequenziamento PCR è stata la seguente: NDM 67,6%, lieviti 26%, dermatofiti 4,8% e infezioni miste 1,6%.
| Lo studio di coorte retrospettivo comprendeva campioni di unghie di pazienti che si erano rivolti al laboratorio specializzato in dermatomicologia del reparto di dermatologia dell’Ospedale Universitario di Losanna (CHUV) nel 2013-2018 a causa di un sospetto clinico di onicomicosi. In tutti i casi in cui la diagnostica micologica convenzionale non ha prodotto risultati chiari, è stato effettuato il sequenziamento PCR. secondo [1] |
Letteratura:
- Pospischil I, et al.: Identificazione degli agenti dermatofiti e non dermatofiti nell’onicomicosi mediante PCR e sequenziamento del DNA – un confronto retrospettivo degli strumenti diagnostici. Journal of Fungi 2022; 8(10): 1019.
- Sigurgeirsson B, Baran R: La prevalenza dell’onicomicosi nella popolazione globale – Uno studio della letteratura. JEADV 2014; 28: 1480-1491.
- Verrier J, Monod M: Diagnosi della dermatofitosi con la biologia molecolare. Micopatologia 2017; 182: 193-202.
- Petinataud D, et al: Diagnosi molecolare dell’onicomicosi. J Mycol Med 2014; 24: 287-295.
PRATICA DERMATOLOGICA 2023; 33(1): 39