Milioni di persone in tutto il mondo soffrono di malattie infiammatorie intestinali (IBD) e la prevalenza è in aumento. Per saperne di più sul ruolo del microbioma, i ricercatori del Massachusetts General Hospital hanno analizzato i dati sul microbiota intestinale dei pazienti con IBD e dei controlli sani. Hanno scoperto che alcuni ceppi di batteri intestinali sono associati alle IBD. I risultati sono stati pubblicati sulla rivista Cell Host & Microbe.
La malattia di Crohn (morbo di Crohn) e la colite ulcerosa (colite ulcerosa), i due sottotipi di IBD, sono diversi a livello clinico e molecolare, ma entrambi condividono prove convincenti di un’eziologia basata sul microbioma [1,2]. “L’infiammazione esercita un’enorme pressione selettiva sui batteri intestinali, quindi abbiamo ipotizzato che il microbioma intestinale possa contenere ceppi batterici unici che sono associati ai processi patologici infiammatori in termini di abbondanza e adattamento genetico”, ha spiegato il primo autore, il dottor Adarsh Kumbhari, ricercatore in Medicina presso il Massachusetts General Hospital [3]. “Per verificare questa ipotesi, stiamo utilizzando approcci evolutivi per individuare i ceppi batterici nel contesto delle IBD, ossia la malattia di Crohn e la colite ulcerosa”. In particolare, si tratta del metagenoma fecale, che consente analisi su larga scala per mappare i ceppi batterici alla base dei processi infiammatori [1]. Il dottor Kumbhari ha spiegato che l’identificazione di questi ceppi batterici può aiutare a scoprire quali strategie molecolari utilizzano i batteri per sopravvivere durante i processi infiammatori e a scoprire di più sulle nuove interazioni microbioma-ospite che sono rilevanti per il rischio di malattia [3].
Analisi in più fasi dei campioni di feci
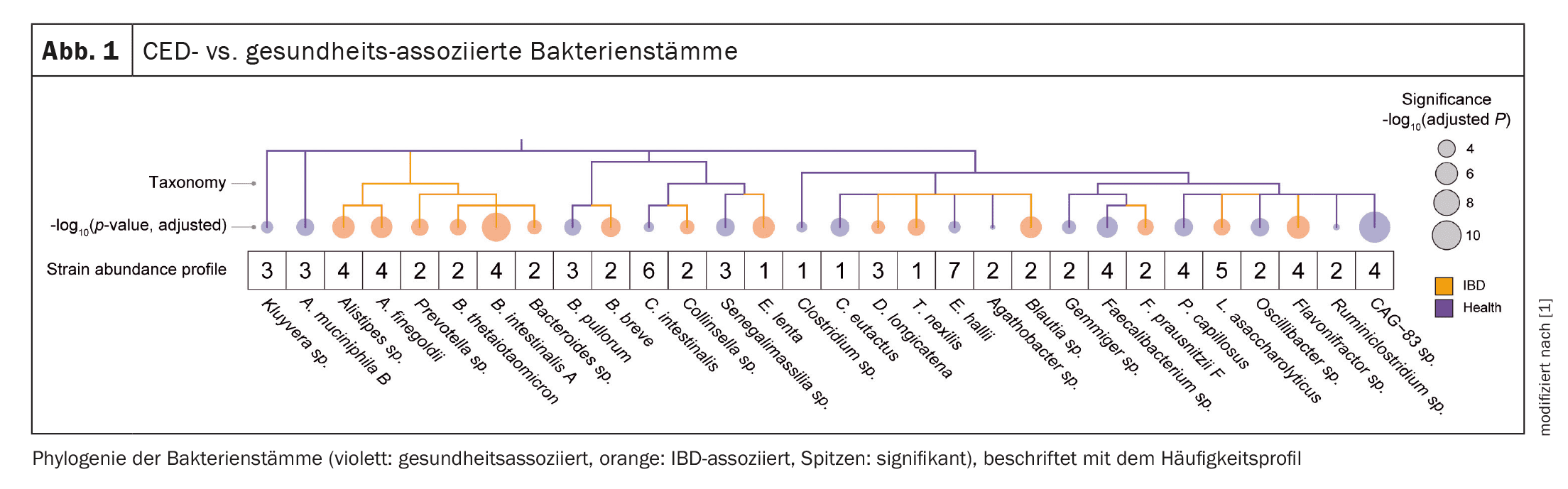
Basandosi sui set di dati di 11 studi condotti tra il 2009 e il 2021, il team di ricerca ha estratto i dati dei campioni di feci di 822 pazienti IBD e 1257 controlli sani, concentrandosi sui genotipi dei ceppi batterici [1,3]. Ciò ha rivelato che centinaia di ceppi batterici erano più diffusi nei campioni di feci dei pazienti con IBD rispetto ai controlli sani e che questi ceppi mostravano relazioni evolutive a lungo termine con la malattia. L’analisi genomica di questi ceppi ha rivelato differenze chiave nei percorsi metabolici legati alla malattia, tra cui lo stress ossidativo, la biosintesi di nutrienti essenziali, la motilità e la parete cellulare batterica [1].
In una fase successiva, hanno analizzato i campioni di feci dei singoli pazienti con IBD nel corso del tempo e hanno scoperto che i ceppi batterici associati all’IBD predominavano durante i periodi di grave infiammazione rispetto ai controlli sani, il che implica che le innovazioni genetiche avevano dato loro un vantaggio di sopravvivenza nel contesto dell’IBD [3]. Le differenze genetiche tra i ceppi batterici associati all’IBD e quelli associati alla salute sono state mappate in relazione agli aspetti noti dell’infiammazione, tra cui lo stress ossidativo, la sintesi dei nutrienti e l’invasione del sistema immunitario.
La Figura 1 mostra una panoramica di alcuni ceppi batterici associati alla CED rispetto a quelli associati alla salute, che i ricercatori hanno identificato in base all’analisi dei cluster precedenti su diversi campioni di feci.
Correlazione con la calprotectina fecale
I ceppi associati alla malattia sono stati trovati anche in individui sani, ma non riflettono il ceppo dominante. Questi potrebbero fornire una resistenza alla colonizzazione, con ceppi commensali associati all’IBD che occupano nicchie infiammatorie per prevenire la colonizzazione opportunistica da parte di altri invasori batterici [1]. “Abbiamo anche scoperto che la perdita di ceppi batterici associati alla salute prevedeva concentrazioni fecali più elevate di calprotectina – un marcatore dell’infiammazione”, ha detto il dottor Christopher S. Smillie, ricercatore senior presso il Center for Computational and Integrative Biology del Massachusetts General [3]. “I nostri risultati possono avere un’utilità diagnostica e il potenziale per contribuire allo sviluppo di interventi mirati per l’IBD e altre malattie immunomediate”, ha spiegato l’esperto [1].
Letteratura:
- Kumbhari A, et al.: Discovery of disease-adapted bacterial lineages in inflammatory bowel diseases. Cell Host & Microbe 2024; Volume 32: 1147–1162.
- Schirmer M, et al.: Microbial genes and pathways in inflammatory bowel disease. Nat Rev Microbiol 2019; 17: 497–511.
- «Research Identifies Gut Bacterial Strains Linked To Inflammatory Bowel Disease», Massachusetts General Hospital, 24.06.2024.
HAUSARZT PRAXIS 2024; 19(11): 30