Come si possono usare gli inibitori della chinasi in modo sensato? Quali sono i vantaggi o gli svantaggi di una combinazione? In che modo il sequenziamento genomico può contribuire a un approccio terapeutico individualizzato con tali inibitori e quali disegni di studio dovrebbero essere utilizzati per verificarlo? Cosa si può imparare dagli studi falliti e dai processi di sviluppo degli inibitori della chinasi conosciuti? Tutte queste domande sono state discusse al Congresso ESMO di Amsterdam.
Annette K. Larsen, Parigi, ha posto la questione se e come combinare gli inibitori della chinasi:
“Lo sviluppo di nuovi agenti antitumorali che mirano alle vie di segnalazione oncogenica rappresenta un’importante svolta concettuale. Tuttavia, i risultati clinici sono stati spesso inferiori alle aspettative, in parte a causa di mutazioni a valle, cicli di feedback inaspettati o il cosiddetto cross-talk delle vie di segnalazione”.
Per questo motivo, oggi c’è una forte attenzione nel puntare su diversi percorsi del segnale simultaneamente o su diverse fasi dello stesso percorso del segnale. Spesso, gli inibitori della chinasi sono stati aggiunti direttamente agli agenti citotossici consolidati, senza aggiustare la dose, causando talvolta gravi effetti collaterali tossici. Nella sua presentazione, Larsen ha delineato le possibilità e i limiti delle combinazioni di inibitori della chinasi.
Due recenti studi di fase III (PACCE e CAIRO2) hanno valutato l’aggiunta di anticorpi monoclonali (mAbs) mirati all’EGFR, rispettivamente cetuximab e panitumumab, a bevacizumab più chemioterapia nei pazienti con carcinoma colorettale (CRC). Questa combinazione non solo era associata a una minore sopravvivenza libera da progressione, ma anche a una peggiore qualità di vita, anche nei pazienti con un tumore KRAS di tipo selvaggio.
“Perché la combinazione dei due mAb non ha funzionato? In primo luogo, la combinazione dei farmaci target era troppo tossica e, in secondo luogo, non è attiva, cioè entrambi inibiscono i ligandi extracellulari o i recettori, ma hanno effetti limitati, forse addirittura nulli, sulla segnalazione della tirosin-chinasi recettoriale (RTK)”, afferma Larsen. Conclude che, sebbene la combinazione di agenti mirati a VEGF ed EGFR sia possibile, non deve necessariamente essere utilizzata contemporaneamente alla chemioterapia. Lo stato di mutazione KRAS svolge un ruolo importante, soprattutto nei mAb mirati all’EGFR.
Approcci terapeutici individualizzati
Il Prof. Dr. med. Emile E. Voest, di Utrecht, ha parlato delle possibilità di includere il sequenziamento genomico nel processo decisionale terapeutico: “Informazioni dettagliate su come si forma geneticamente un tumore consentono di selezionare in modo più mirato pazienti specifici per una rispettiva terapia. Esempi come il trastuzumab per l’espressione di HER2 nel tumore al seno, l’imatinib per le traslocazioni BCR-ABL nella leucemia, il vemurafenib e il crizotinib contro le mutazioni V600E nel melanoma o le traslocazioni ALK-EML4 nel tumore al polmone hanno dimostrato chiaramente la validità del concetto. Questi successi spettacolari sono rovinati dal fatto che sono temporanei, perché spesso si sviluppa una resistenza”.
Grazie ai progressi della tecnologia di sequenziamento, oggi è possibile generare informazioni dettagliate sulle anomalie genomiche di un tumore. Le analisi di un singolo gene (ad esempio, BRAF, KRAS) saranno sostituite da analisi dell’intero genoma nel prossimo futuro. Questo approccio globale consente approfondimenti a livello di vie di segnalazione, piuttosto che a livello di singoli geni. “Per esempio, non siamo riusciti a trovare una correlazione tra il successo della chemioterapia e le mutazioni specificamente rintracciabili nel gene PI3K nelle pazienti con cancro al seno, ma molto una con le mutazioni nella via di segnalazione PI3K”, dice il Prof. Voest.
L’eterogeneità tumorale è una dimensione importante della crescita del tumore e la crescita clonale di popolazioni resistenti si verifica spesso durante il trattamento. In questo caso, i metodi di “sequenziamento (ultra)profondo” possono aiutare a rilevare e identificare precocemente i cloni, consentendo un approccio terapeutico anticipato. L’opinione attuale è che sarà più facile trovare un profilo genetico predittivo per i farmaci con un meccanismo d’azione specifico (come il vemurafenib) che per gli inibitori della chinasi ad ampio spettro (come il sunitinib). Tenendo conto delle scoperte sulla suddetta correlazione tra il successo della chemioterapia e la via di segnalazione PI3K, questo approccio può essere considerato un equivoco.
“Siamo all’inizio di un’era in cui i test genetici approfonditi del tumore e del DNA germinale faranno parte della diagnostica regolare dei pazienti oncologici. In particolare, l’interpretazione competente dei dati estremamente complessi sarà fondamentale per la scelta corretta della terapia guidata dal DNA. Per questo, sono necessari grandi database che offrano la possibilità di collegare i successi clinici con i dati genetici”, ha concluso il Prof. Voest.
Studi sul genotipo vs. studi sul paniere
“Gli studi sul genotipo si basano sull’analisi di un certo numero di pazienti affetti da una malattia per verificarne il genotipo e poi assegnarli a diversi farmaci mirati in base al loro profilo”, afferma José Baselga, MD, New York (Fig. 1). “I problemi di questo approccio sono i seguenti: I farmaci utilizzati in ciascun gruppo di solito non sono i migliori della categoria, ma sono disponibili (ad esempio, il primo studio BATTLE ha utilizzato sorafenib come inibitore di RAF). Inoltre, se si utilizza la randomizzazione, nel corso dello studio potrebbe sembrare non etico randomizzare alcuni pazienti che non saranno trattati in base al loro profilo. Inoltre, il numero totale di partecipanti è di solito basso, per cui questo disegno può spesso acquisire un numero troppo basso di pazienti con mutazioni rare per confrontare validamente l’esito clinico con il profilo genetico (ad esempio BRAF nel cancro del polmone o mutazioni ERBB2 nel cancro ovarico)”.
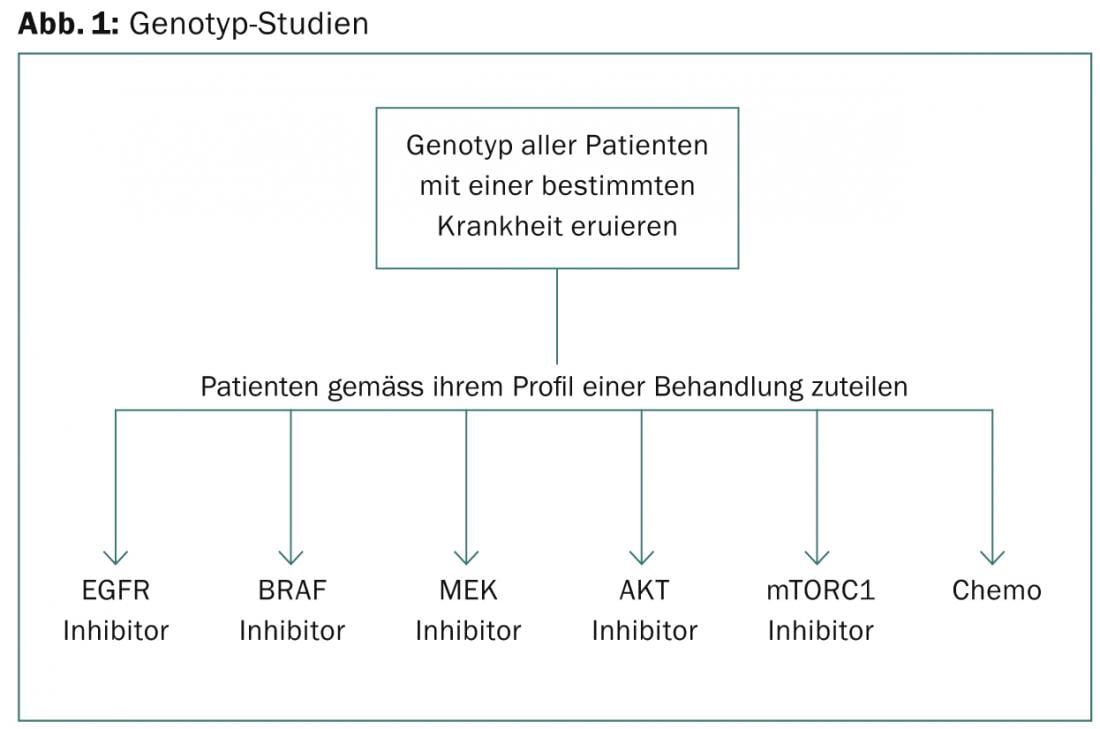
Così si chiama. Gli studi sui cestini, invece, consentono di verificare un’ipotesi concreta, ad esempio: I pazienti con cancro biliare il cui tumore presenta mutazioni BRAF(V600E) rispondono a vemurafenib? Inoltre, poiché questo tipo di studio si basa su una raccolta di coorti specifiche per la malattia, può essere utilizzato per indagare l’influenza dell’ascendenza sulla risposta ai farmaci. Inoltre, il campionamento dei tessuti può spiegare l’eterogeneità della risposta. La critica principale agli studi a paniere è che possono perdere quei pazienti che potrebbero rispondere, ma che non hanno il biomarcatore che stanno cercando. Inoltre, l’identificazione dei partecipanti rimane un ostacolo: secondo Baselga, è quindi indispensabile separare il protocollo di screening dal protocollo di trattamento.
Imparare dagli errori
Come possiamo imparare dagli studi falliti sugli inibitori della chinasi? Questa domanda è stata posta da Stefan Sleijfer, MD, Rotterdam: “Negli ultimi anni, diversi nuovi inibitori della chinasi sono stati approvati rapidamente e con successo, grazie ai risultati di studi in parte sensazionali. Al contrario, però, molti di questi agenti hanno fallito, a volte già nella fase clinica iniziale, a volte solo dopo aver condotto studi di fase III ampi e costosi. Sia dai processi di sviluppo riusciti che da quelli falliti si possono trarre conclusioni preziose per i futuri progetti di studio”.
Gli inibitori della chinasi di maggior successo sono quelli che mirano e inibiscono direttamente il prodotto di un gene mutato, cioè colpiscono un sottogruppo specifico di una malattia. Una volta conosciuto il meccanismo d’azione di questi farmaci e quindi potendo selezionare in modo specifico i pazienti idonei, i principi attivi sono stati testati clinicamente in gruppi di pazienti specificamente selezionati fin dall’inizio. Esempi di successo sono l’imatinib per i tumori stromali gastrointestinali maligni (GIST), il vemurafenib per il melanoma mutato BRAF(V600E) o il crizotinib per il tumore polmonare non a piccole cellule (NSCLC) con gene di fusione EML4-ALK.
Un’importante caratteristica comune di quei farmaci che hanno fallito o ottenuto l’approvazione solo dopo un percorso arduo e costoso è che il meccanismo d’azione era sconosciuto prima dell’inizio degli studi clinici. Pertanto, non è stato possibile stabilire profili predittivi, non è stato possibile identificare a priori i pazienti suscettibili di rispondere e gli studi clinici sono stati condotti su popolazioni di pazienti non selezionate. Gli esempi attuali sono gli antagonisti dell’IGF-1R e gli inibitori di mTOR.
“Quindi la lezione più importante di questi studi è il vantaggio e la necessità di scoprire l’esatto meccanismo d’azione del farmaco in esame in ambito preclinico e di identificare i marcatori predittivi prima di avviare grandi studi clinici. Se il meccanismo è sconosciuto, rimane discutibile se il farmaco in questione debba entrare nel processo di sviluppo. Soprattutto perché questo richiede una quantità estremamente elevata di risorse e di denaro che potrebbero essere meglio spesi per altri composti da testare. Se tuttavia un farmaco con un meccanismo d’azione sconosciuto è già in fase di sviluppo, si possono utilizzare i disegni adattivi. Ad esempio, la raccolta di materiale biologico è essenziale per poter scoprire retrospettivamente i profili predittivi, come nel caso degli inibitori della chinasi EGFR nel NSCLC”, ha riassunto il dottor Sleijfer.
Fonte: “Uso ottimale degli inibitori mirati delle chinasi (TKI)”, Simposio al Congresso ESMO, 27 settembre – 1 ottobre 2013, Amsterdam.
InFo ONCOLOGIA & EMATOLOGIA 2014; 2(1): 45-47