Milhões de pessoas em todo o mundo sofrem de doença inflamatória intestinal (DII) e a sua prevalência está a aumentar. Para saber mais sobre o papel do microbioma, investigadores do Massachusetts General Hospital analisaram dados sobre o microbiota intestinal de doentes com DII e de controlos saudáveis. Descobriram que certas estirpes de bactérias intestinais estão associadas à DII. Os resultados foram publicados na revista Cell Host & Microbe.
[1,2]A doença de Crohn (doença de Crohn) e a colite ulcerosa (colite ulcerosa), os dois subtipos de DII, são diferentes a nível clínico e molecular, mas ambas partilham provas convincentes de uma etiologia baseada no microbioma . “A inflamação exerce uma enorme pressão selectiva sobre as bactérias intestinais, pelo que levantámos a hipótese de o microbioma intestinal poder conter estirpes bacterianas únicas que estão associadas a processos de doenças inflamatórias em termos de abundância e adaptação genética”, explicou o primeiro autor, Dr. Adarsh Kumbhari, investigador em medicina no Massachusetts General Hospital [3]. “Para testar esta hipótese, estamos a utilizar abordagens evolutivas para detetar estirpes bacterianas no contexto das DII, ou seja, a doença de Crohn e a colite ulcerosa”. Especificamente, isto envolve o metagenoma fecal, que permite análises em grande escala para mapear as estirpes bacterianas que estão na base dos processos inflamatórios [1]. O Dr. Kumbhari explicou que a identificação destas estirpes bacterianas pode ajudar a descobrir quais as estratégias moleculares que as bactérias utilizam para sobreviver durante os processos inflamatórios e a descobrir mais sobre as novas interações microbioma-hospedeiro que são relevantes para o risco de doença [3].Análise em várias fases de amostras de fezes
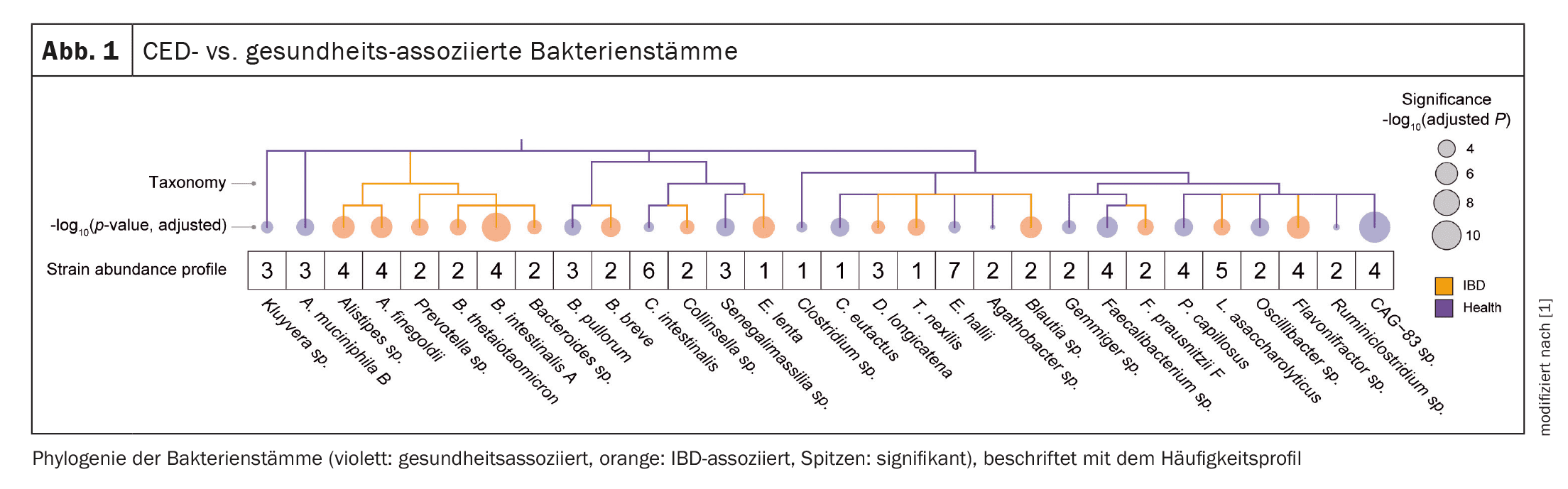
[1,3]Com base em conjuntos de dados de 11 estudos realizados entre 2009 e 2021, a equipa de investigação extraiu dados de amostras de fezes de 822 doentes com DII e 1257 controlos saudáveis, centrando-se nos genótipos das estirpes bacterianas . Isto revelou que centenas de estirpes bacterianas eram mais prevalentes em amostras de fezes de doentes com DII do que em controlos saudáveis e que estas estirpes apresentavam relações evolutivas a longo prazo com a doença. A análise genómica destas estirpes revelou diferenças fundamentais nas vias metabólicas relacionadas com a doença, incluindo o stress oxidativo, a biossíntese de nutrientes essenciais, a motilidade e a parede celular bacteriana [1].Numa fase seguinte, analisaram as amostras de fezes de doentes com DII ao longo do tempo e descobriram que as estirpes bacterianas associadas à DII predominavam durante os períodos de inflamação grave em comparação com os controlos saudáveis, o que implica que as inovações genéticas lhes tinham dado uma vantagem de sobrevivência no contexto da DII [3]. As diferenças genéticas entre as estirpes bacterianas associadas à DII e as associadas à saúde foram mapeadas em relação a aspectos conhecidos da inflamação – incluindo o stress oxidativo, a síntese de nutrientes e a invasão do sistema imunitário.
A Figura 1 mostra uma visão geral de algumas estirpes bacterianas associadas a CED versus estirpes associadas à saúde que os investigadores identificaram com base numa análise prévia de grupos em diferentes amostras de fezes.
Correlação com a calprotectina fecal
As estirpes associadas à doença também foram encontradas em indivíduos saudáveis, mas não reflectiram a estirpe dominante. Estas poderiam proporcionar resistência à colonização, com estirpes de bactérias comensais associadas à DII a ocuparem nichos inflamatórios para evitar a colonização oportunista por outros invasores bacterianos [1]. “Descobrimos também que a perda de estirpes bacterianas associadas à saúde previa concentrações fecais mais elevadas de calprotectina – um marcador de inflamação”, afirmou o Dr. Christopher S. Smillie, investigador sénior do Center for Computational and Integrative Biology do Massachusetts General [3]. “As nossas descobertas podem ter utilidade diagnóstica e o potencial de contribuir para o desenvolvimento de intervenções direcionadas para a DII e outras doenças imunomediadas”, explicou o especialista [1].
Literatura:
- Kumbhari A, et al: Descoberta de linhagens bacterianas adaptadas a doenças em doenças inflamatórias intestinais. Cell Host & Microbe 2024; Volume 32: 1147-1162.
- Schirmer M, et al: Genes e vias microbianas na doença inflamatória intestinal. Nat Rev Microbiol 2019; 17: 497-511.
- “Research Identifies Gut Bacterial Strains Linked To Inflammatory Bowel Disease”, Massachusetts General Hospital, 24.06.2024.
PRÁTICA DE CLÍNICA GERAL 2024; 19(11): 30