En el diagnóstico de tumores, el término biopsias líquidas hace referencia al análisis del ADN circulante libre de células (cfADN) y de las células tumorales circulantes. Hoy en día es posible detectar las mutaciones de un tumor avanzado de forma mínimamente invasiva mediante análisis de cfADN. Para las pruebas de cfADN debe utilizarse plasma, no suero. Las células tumorales circulantes podrán utilizarse en un futuro próximo para el análisis de marcadores predictivos, como para la determinación de la variante de empalme AR-V7 en el carcinoma de próstata resistente a la castración.
Aunque el término biopsias líquidas es relativamente nuevo en el contexto del diagnóstico molecular de tumores, se ha aceptado ampliamente entre patólogos y oncólogos. En el diagnóstico de tumores, este término se utilizó primero para el análisis de células tumorales circulantes (CTC) y sólo más tarde para el ADN libre de células circulantes (cfADN). En resumen, se trata de un término genérico para referirse a la extracción de material de un tumor sólido mediante la simple toma de una muestra de sangre. No obstante, el término “biopsia líquida” no se limita exclusivamente a su uso en el diagnóstico de tumores: En el diagnóstico prenatal, las muestras de sangre pueden utilizarse para extraer conclusiones sobre las anomalías cromosómicas del feto.
Interés por la detección mínimamente invasiva
Las células tumorales circulantes (CTC) y el ADN libre de células circulante (cfADN) se han investigado durante décadas y se han debatido como posibles marcadores de diagnóstico y pronóstico en la literatura. La primera descripción del CTC tuvo lugar en el siglo XIX por el australiano J. Ashworth (1869), la del cfADN algo menos de un siglo después por los dos franceses P. Mandel y P. Metais (1948).
Las CTC pueden originarse tanto en el tumor primario como en las metástasis. Aún no está claro si la liberación de CTC es un proceso activo y, por tanto, parte de un programa biológico de un tumor, o si la liberación se produce de forma aleatoria. Sin embargo, se sabe que algunas de estas células tienen la capacidad de sobrevivir en el torrente sanguíneo y seguir multiplicándose en otras partes del cuerpo humano. para formar una metástasis. Se discute controvertidamente si las células individuales son suficientes para ello o si las asociaciones (clusters) de células tumorales no son responsables del proceso de metástasis a distancia hematógena [1].
El ADN circulante libre de células (cfADN), en cambio, procede de células moribundas (apoptóticas o necróticas). El cfADN está formado por ADN procedente tanto de células tumorales (ADN tumoral circulante libre de células, abreviado ctADN) como de células sanas (ADN normal circulante libre de células, abreviado cnADN). Es controvertido si el cfADN de la sangre cumple alguna función. En comparación con el CTC (1-2,5 horas), el cfADN tiene una semivida relativamente corta de 15-30 minutos.
Tanto las CTC como el cfADN/ADNct están adquiriendo una gran importancia para el diagnóstico de tumores gracias a las nuevas posibilidades de investigación genómica. Hoy en día, la secuenciación de nueva generación (NGS) permite secuenciar genomas enteros en muy poco tiempo. Desde el uso de esta tecnología en un gran número de ensayos clínicos, el número de marcadores genómicos potencialmente predictivos ha aumentado considerablemente y, en consecuencia, el interés por la detección mínimamente invasiva de estos marcadores predictivos en la sangre.
Determinar las mutaciones de forma significativa
En comparación con la secuenciación Sanger convencional, la NGS permite secuenciar regiones más amplias simultáneamente y, sobre todo, con una gran profundidad (redundancia). Esta profundidad es necesaria si se quieren detectar mutaciones subclonales o si el ADN tumoral representa sólo una fracción del ADN total que se va a analizar. Este último es el caso del análisis del ctADN: dependiendo de la entidad tumoral y del estadio de la enfermedad tumoral, el contenido de ctADN en la sangre puede variar mucho o ascender hasta unos pocos por mil.
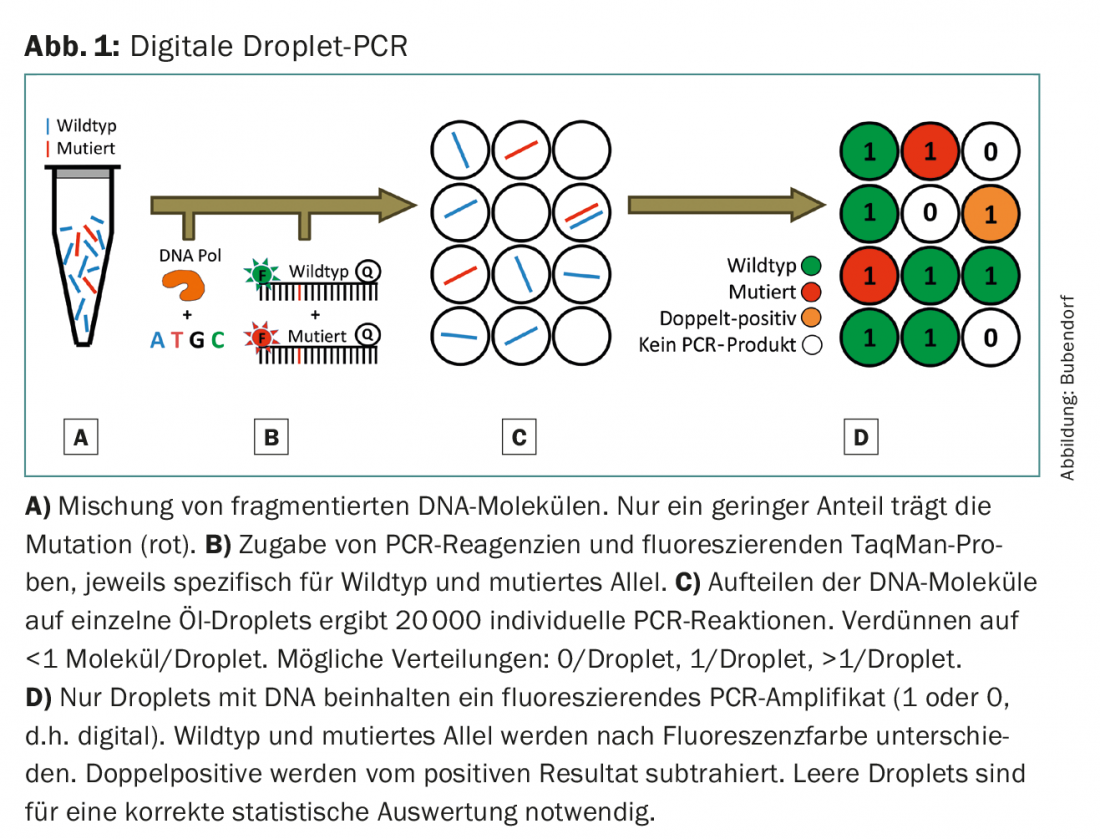
Con la mayoría de los secuenciadores NGS, los protocolos estándar pueden detectar mutaciones que sólo representan el 2-5% del ADN total. Sin embargo, el ctADN suele estar por debajo de este umbral, especialmente en tumores aún no avanzados o en pacientes sometidos a terapia. A diferencia de la NGS, la PCR digital permite determinar una mutación en este rango de alta sensibilidad: con la ayuda de sondas específicas marcadas con fluorescencia, se pueden determinar de forma significativa ciertas mutaciones en el rango del 0,01%. El procedimiento y la técnica se describen utilizando el ejemplo de la PCR digital en gotitas (Fig. 1) . La desventaja de este método es que debe llevarse a cabo una reacción distinta para cada mutación que se quiera determinar. Por lo tanto, no se recomienda utilizar esta metodología para una investigación general del genoma, sino sólo para la investigación de mutaciones aisladas.
Células tumorales circulantes (CTC)
En la última década, varios estudios han demostrado que la detección de una mayor cantidad de CTC en la sangre de pacientes con cáncer avanzado se asocia a una mala supervivencia [2]. En este contexto, la autoridad reguladora estadounidense FDA ha aprobado la recogida del número de CTC para la evaluación del pronóstico en pacientes con cáncer metastásico de próstata, colorrectal y de mama mediante el dispositivo CellSearch® (Veridex). Se trata del único dispositivo aprobado por la FDA para la determinación de CTC.
La detección o el enriquecimiento de células tumorales circulantes sigue siendo un reto tecnológico: En 10 ml de sangre total hay unos 50.000 millones de glóbulos rojos, 50 millones de glóbulos blancos, pero sólo entre 0 y 100 CTC. Básicamente, existen dos métodos técnicos para detectar y registrar CTC en la sangre. Para ello se puede utilizar un enfoque basado en las propiedades biológicas de las CTC, en particular en la expresión de proteínas que sólo se encuentran en las CTC pero no en las células sanguíneas. Así, las CTC pueden capturarse con anticuerpos contra proteínas específicas de las células epiteliales como la EpCAM o las citoqueratinas. Otra posibilidad es reconocer las CTC en función de sus diferentes propiedades físicas, como la densidad, el tamaño o la deformabilidad.
Diagnóstico predictivo de tumores con CTC para el futuro
En el pasado, los ensayos clínicos se centraban en el número de CTC y su correlación con la supervivencia. Gracias a los avances tecnológicos, ahora es posible pescar estas células y analizarlas mediante NGS u otras tecnologías, como la PCR en tiempo real [3]. Aunque en la actualidad este enfoque se utiliza principalmente en investigación, existen fundadas esperanzas de que en el futuro puedan realizarse diagnósticos tumorales predictivos con estas células.
El ejemplo más prometedor de uso predictivo de CTC hasta la fecha es la determinación del estado AR-V7 en pacientes con cáncer de próstata resistente a la castración. El AR-V7 es una variante de empalme del gen del receptor de andrógenos que da lugar a una proteína alterada que es constitutivamente activa. Antonarakis et al. pudieron demostrar que los pacientes cuyas CTC expresaban el AR-V7 ARN respondían peor a las terapias con enzalutamida y abiraterona [4]. El ensayo utilizado en este estudio se basa en capturar las CTC mediante anticuerpos y realizar la PCR en tiempo real con cebadores específicos AR-V7 sobre el ARNm extraído. Esta prueba AR-V7 basada en CTC se comercializará en un futuro próximo. Podría considerarse un hito en el uso de CTC para detectar mutaciones, fusiones de genes y otros marcadores predictivos.
ADN circulante libre de células (cfADN)
Mientras que los primeros estudios de cfADN investigaron una posible correlación de la cantidad de cfADN con el pronóstico, hoy en día la NGS puede utilizarse para determinar la secuencia de bases del cfADN (es decir, el ctADN) procedente de las células tumorales. Dado que la diferenciación entre el cfADN general y el ctADN no es posible por razones técnicas al extraer el cfADN, la mayor parte del cfADN puede consistir en cnADN y no en ctADN. Para poder seguir determinando las mutaciones en el ctADN, debe alcanzarse una gran profundidad con la NGS. Se requiere plasma sanguíneo para las pruebas de cfADN.
Gracias a la NGS, recientemente ha sido posible detectar las mutaciones del tumor en el ADNct de hasta el 75% de los pacientes con tumores avanzados [5]. En los tumores no avanzados, la tasa de detección desciende hasta aproximadamente el 50%. Sin embargo, cabe suponer que estos índices aumentarán gracias al uso de métodos más sensibles.
Varios estudios han demostrado que la cantidad de ctADN se correlaciona con la carga tumoral: si el tumor responde a la terapia, la cantidad de ctADN en la sangre del paciente disminuye. Lo contrario ocurre con la aparición de una recidiva: en un pequeño grupo de pacientes con cáncer de mama, Dawson et al. muestran un aumento de los niveles de ctADN en sangre semanas antes de que el diagnóstico por imagen estándar pueda detectar la recidiva [6]. Para estos exámenes, basta con determinar o cuantificar algunas mutaciones en la sangre específicas del tumor correspondiente. Si se quiere utilizar la biopsia líquida para investigar si el tumor de un paciente tiene una mutación predictiva, se suelen utilizar los llamados paneles para secuenciación, que varían mucho en tamaño (Fig. 2).
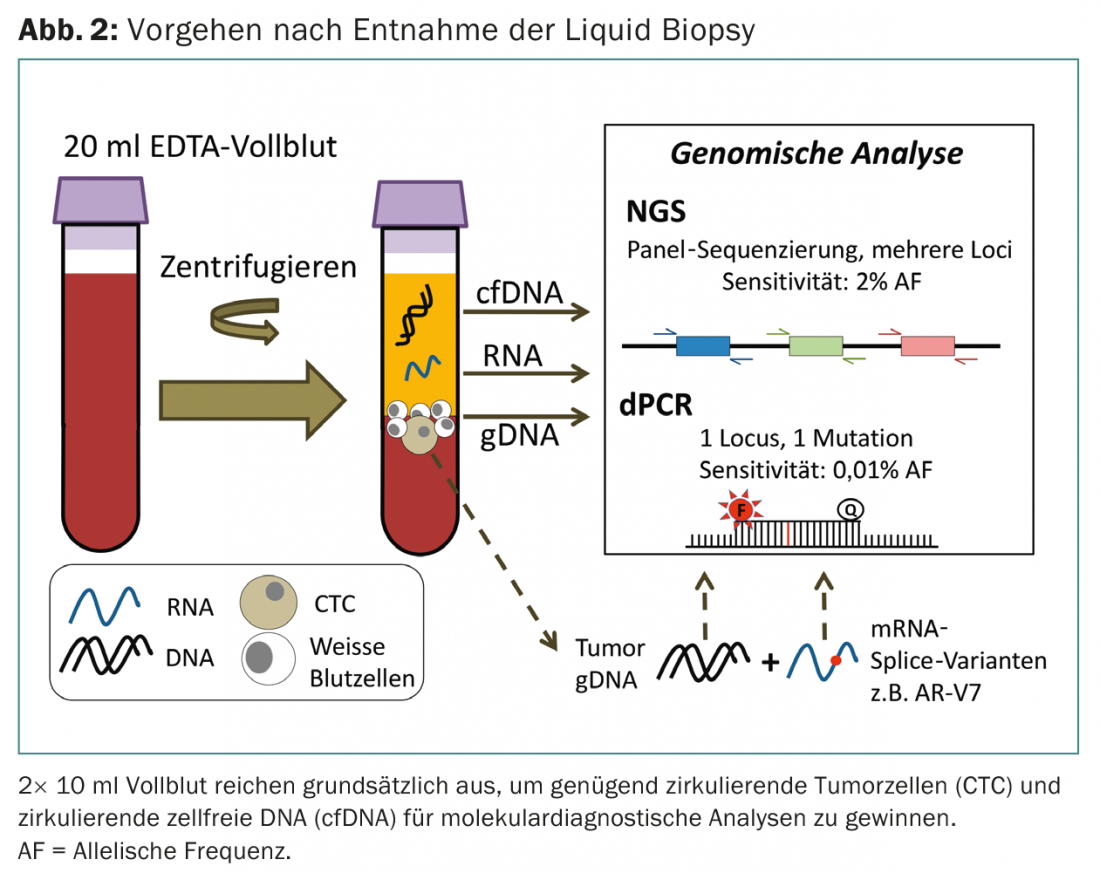
Los paneles estándar utilizados en el diagnóstico de tumores abarcan mutaciones de entre 20 y 50 genes. Estos genes son genes relevantes para la terapia como EGFR, KRAS, NRAS o BRAF. Dado que, como se ha mencionado, para la secuenciación del cfADN se requieren profundidades de secuenciación elevadas, los recursos y los costes de secuenciación aumentan con el tamaño del panel.
Debido a la gran relevancia clínica de las aplicaciones, los exámenes basados en el cfADN están ya muy cerca de la práctica clínica diaria: el departamento de patología del Hospital Universitario de Basilea ofrece análisis de cfADN para el diagnóstico desde abril de este año. Esto implica el uso de paneles NGS o PCR digital para examinar el cfADN de los pacientes en busca de mutaciones con propiedades predictivas. Esto es de especial interés si una biopsia de tejido no es posible o no es razonable para un paciente. Esta aplicación se está introduciendo actualmente sobre todo en el diagnóstico predictivo de los carcinomas de pulmón. Sin embargo, es previsible una expansión a otras entidades tumorales.
Mediante los exámenes de cfADN pueden determinarse de forma no invasiva las mutaciones del EGFR conocidas y relevantes para la terapia, en particular también la mutación T790M del EGFR, que es predictiva de la respuesta al osimertinib en pacientes con resistencia a los inhibidores de la tirosina quinasa del EGFR (EGFR-TKI).
Un caso práctico
Hace tres años, se detectó una mutación en el gen EGFR (p.E746-T751delinsF) en un paciente con un adenocarcinoma de pulmón TTF1-positivo en estadio IV mediante la secuenciación de células tumorales procedentes de un lavado broncoalveolar. La paciente fue entonces tratada con el EGFR TKI erlotinib durante ocho meses y posteriormente, como terapia de segunda línea, con afatinib (Fig. 3).
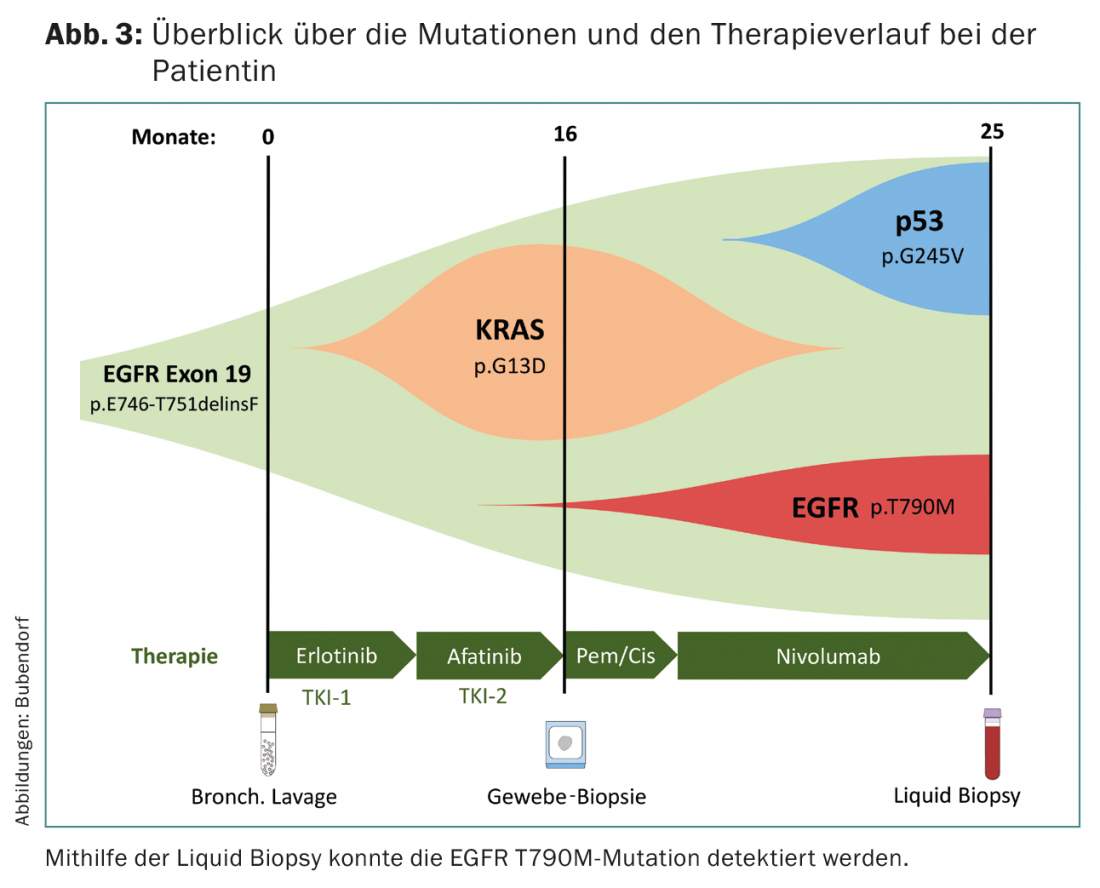
La secuenciación de una biopsia de tejido reveló una mutación KRAS (p.G13D) sugestiva de resistencia a los EGFR-TKI. La paciente fue cambiada a quimioterapia de corta duración debido a los graves efectos secundarios de los TKI del EGFR, seguida de tratamiento con el inhibidor de la PD 1 nivolumab. El diagnóstico por imagen mostró progresión tumoral en ese momento. Debido a la inaccesibilidad del tumor, se realizó una biopsia líquida: Además de la mutación original del EGFR, se detectaron de nuevo una mutación TP53 y una mutación EGFR T790M. Este último es predictivo de la respuesta al osimertinib. La paciente fue tratada con osimertinib y mostró una respuesta parcial en la tomografía computarizada dos meses después de iniciar la terapia.
Literatura:
- Cheung KJ, et al.: Una ruta colectiva hacia la metástasis: la siembra por grupos de células tumorales. Science 2016 abr 8; 352(6282): 167-169.
- De Bono JS, et al: Las células tumorales circulantes predicen el beneficio de supervivencia del tratamiento en el cáncer de próstata metastásico resistente a la castración. Clin Cancer Res 2008 Oct 1; 14(19): 6302-6309.
- Lohr JG, et al: La secuenciación del exoma completo de las células tumorales circulantes proporciona una ventana al cáncer de próstata metastásico. Nat Biotechnol 2014 mayo; 32(5): 479-484.
- Antonarakis ES, et al: AR-V7 y resistencia a la enzalutamida y la abiraterona en el cáncer de próstata. N Engl J Med 2014 Sep 11; 371(11): 1028-1038.
- Bettegowda C, et al: Detección de ADN tumoral circulante en neoplasias humanas en fase temprana y tardía. Sci Transl Med 2014 Feb 19; 6(224): 224ra24.
- Dawson SJ, et al: Análisis del ADN tumoral circulante para monitorizar el cáncer de mama metastásico. N Engl J Med 2013 Mar 28; 368(13): 1199-1209.
InFo ONCOLOGÍA Y HEMATOLOGÍA 2016; 4(4): 10-13