Millones de personas en todo el mundo padecen enfermedad inflamatoria intestinal (EII) y su prevalencia va en aumento. Para averiguar más sobre el papel del microbioma, investigadores del Hospital General de Massachusetts analizaron los datos de la microbiota intestinal de pacientes con EII y de controles sanos. Descubrieron que determinadas cepas de bacterias intestinales están asociadas a la EII. Los resultados se publicaron en la revista Cell Host & Microbe.
[1,2]La enfermedad de Crohn (enfermedad de Crohn) y la colitis ulcerosa (colitis ulcerosa), los dos subtipos de EII, son diferentes a nivel clínico y molecular, pero ambas comparten pruebas convincentes de una etiología basada en el microbioma . “La inflamación ejerce una tremenda presión selectiva sobre las bacterias intestinales, por lo que planteamos la hipótesis de que el microbioma intestinal puede contener cepas bacterianas únicas que se asocian a los procesos inflamatorios de la enfermedad en términos de abundancia y adaptación genética”, explicó el primer autor, el Dr. Adarsh Kumbhari, investigador en Medicina del Hospital General de Massachusetts [3]. “Para probar esta hipótesis, estamos utilizando enfoques evolutivos para detectar cepas bacterianas en el contexto de la EII, es decir, la enfermedad de Crohn y la colitis ulcerosa”. En concreto, se trata del metagenoma fecal, que permite realizar análisis a gran escala para cartografiar las cepas bacterianas que subyacen a los procesos inflamatorios [1]. El Dr. Kumbhari explicó que la identificación de estas cepas bacterianas puede ayudar a descubrir qué estrategias moleculares utilizan las bacterias para sobrevivir durante los procesos inflamatorios y a averiguar más sobre las nuevas interacciones microbioma-huésped que son relevantes para el riesgo de enfermedad [3].Análisis multietapa de muestras de heces
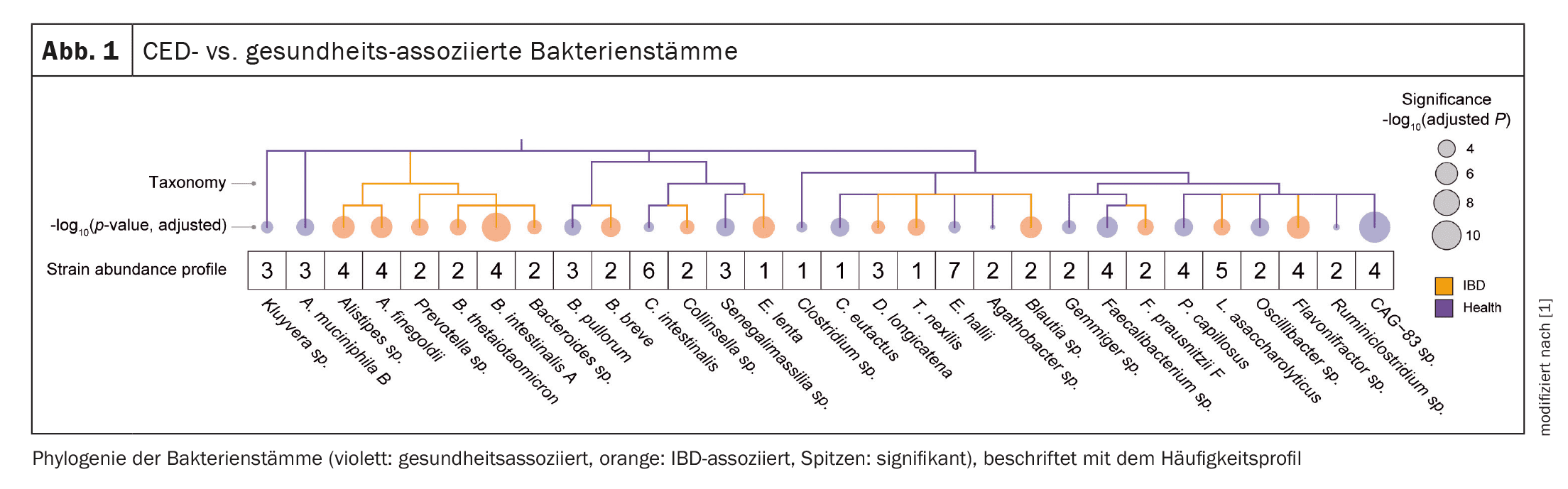
[1,3]Basándose en conjuntos de datos de 11 estudios realizados entre 2009 y 2021, el equipo de investigación extrajo datos de muestras de heces de 822 pacientes con EII y 1257 controles sanos, centrándose en los genotipos de las cepas bacterianas . Esto reveló que cientos de cepas bacterianas eran más prevalentes en las muestras de heces de los pacientes con EII que en los controles sanos y que estas cepas mostraban relaciones evolutivas a largo plazo con la enfermedad. El análisis genómico de estas cepas reveló diferencias clave en las vías metabólicas relacionadas con la enfermedad, como el estrés oxidativo, la biosíntesis de nutrientes esenciales, la motilidad y la pared celular bacteriana [1].En un siguiente paso, analizaron las muestras de heces de pacientes individuales con EII a lo largo del tiempo y descubrieron que las cepas bacterianas asociadas a la EII predominaban durante los periodos de inflamación grave en comparación con los controles sanos, lo que implicaba que las innovaciones genéticas les habían otorgado una ventaja de supervivencia en el contexto de la EII [3]. Se trazaron diferencias genéticas entre las cepas bacterianas asociadas a la EII y las asociadas a la salud en relación con aspectos conocidos de la inflamación, como el estrés oxidativo, la síntesis de nutrientes y la invasión del sistema inmunitario.
La figura 1 muestra un resumen de algunas cepas bacterianas asociadas al CED frente a las asociadas a la salud que los investigadores identificaron basándose en un análisis previo de conglomerados en diferentes muestras de heces.
Correlación con la calprotectina fecal
También se encontraron cepas asociadas a la enfermedad en individuos sanos, pero no reflejaban la cepa dominante. Éstas podrían proporcionar resistencia a la colonización, ya que las cepas comensales asociadas a la EII ocuparían nichos inflamatorios para evitar la colonización oportunista por parte de otros invasores bacterianos [1]. “También descubrimos que la pérdida de cepas bacterianas asociadas a la salud predecía concentraciones fecales más elevadas de calprotectina, un marcador de inflamación”, afirmó el Dr. Christopher S. Smillie, investigador principal del Centro de Biología Computacional e Integrativa del Massachusetts General [3]. “Nuestros hallazgos pueden tener utilidad diagnóstica y el potencial de contribuir al desarrollo de intervenciones específicas para la EII y otras enfermedades inmunomediadas”, explicó el experto [1].
Literatura:
- Kumbhari A, et al.: Discovery of disease-adapted bacterial lineages in inflammatory bowel diseases. Cell Host & Microbe 2024; Volume 32: 1147–1162.
- Schirmer M, et al.: Microbial genes and pathways in inflammatory bowel disease. Nat Rev Microbiol 2019; 17: 497–511.
- «Research Identifies Gut Bacterial Strains Linked To Inflammatory Bowel Disease», Massachusetts General Hospital, 24.06.2024.
HAUSARZT PRAXIS 2024; 19(11): 30